
- Biopython 教程
- Biopython - 首頁
- Biopython - 簡介
- Biopython - 安裝
- 建立簡單的應用程式
- Biopython - 序列
- 高階序列操作
- 序列 I/O 操作
- Biopython - 序列比對
- Biopython - BLAST 概述
- Biopython - Entrez 資料庫
- Biopython - PDB 模組
- Biopython - 基序物件
- Biopython - BioSQL 模組
- Biopython - 群體遺傳學
- Biopython - 基因組分析
- Biopython - 表型微陣列
- Biopython - 繪圖
- Biopython - 聚類分析
- Biopython - 機器學習
- Biopython - 測試技術
- Biopython 資源
- Biopython 快速指南
- Biopython - 有用資源
- Biopython - 討論
Biopython 快速指南
Biopython - 簡介
Biopython 是 Python 最大的、最流行的生物資訊學軟體包。它包含許多用於常見生物資訊學任務的不同子模組。它由 Chapman 和 Chang 開發,主要用 Python 編寫。它還包含 C 程式碼來最佳化軟體的複雜計算部分。它可以在 Windows、Linux、Mac OS X 等作業系統上執行。
基本上,Biopython 是一個 Python 模組的集合,提供處理 DNA、RNA 和蛋白質序列操作的功能,例如 DNA 字串的反向互補、在蛋白質序列中查詢基序等。它提供了許多解析器來讀取所有主要的遺傳資料庫,如 GenBank、SwissPort、FASTA 等,以及執行其他流行的生物資訊學軟體/工具(如 NCBI BLASTN、Entrez 等)的包裝器/介面,在 Python 環境中。它有類似的專案,如 BioPerl、BioJava 和 BioRuby。
特徵
Biopython 可移植、清晰且易於學習語法。下面列出了一些主要功能:
解釋型、互動式和麵向物件。
支援 FASTA、PDB、GenBank、Blast、SCOP、PubMed/Medline、ExPASy 相關的格式。
處理序列格式的選項。
管理蛋白質結構的工具。
BioSQL - 用於儲存序列以及特徵和註釋的標準 SQL 表集。
訪問線上服務和資料庫,包括 NCBI 服務(Blast、Entrez、PubMed)和 ExPASy 服務(SwissProt、Prosite)。
訪問本地服務,包括 Blast、Clustalw、EMBOSS。
目標
Biopython 的目標是透過 Python 語言提供對生物資訊學的簡單、標準和廣泛的訪問。Biopython 的具體目標如下:
提供對生物資訊學資源的標準化訪問。
高質量、可重用的模組和指令碼。
可在叢集程式碼、PDB、樸素貝葉斯和馬爾可夫模型中使用的快速陣列操作。
基因組資料分析。
優勢
Biopython 需要非常少的程式碼,並具有以下優點:
提供用於聚類的微陣列資料型別。
讀取和寫入 Tree-View 型別檔案。
支援用於 PDB 解析、表示和分析的結構資料。
支援 Medline 應用程式中使用的期刊資料。
支援 BioSQL 資料庫,它是所有生物資訊學專案中廣泛使用的標準資料庫。
透過提供模組將生物資訊學檔案解析為特定格式的記錄物件或序列加特徵的通用類來支援解析器開發。
基於食譜風格的清晰文件。
案例研究
讓我們檢查一些用例(群體遺傳學、RNA 結構等),並嘗試瞭解 Biopython 在該領域如何發揮重要作用:
群體遺傳學
群體遺傳學是對群體中遺傳變異的研究,包括檢查和建模基因和等位基因頻率在空間和時間上群體中的變化。
Biopython 提供 Bio.PopGen 模組用於群體遺傳學。此模組包含收集經典群體遺傳學資訊所需的所有必要函式。
RNA 結構
對我們的生命至關重要的三種主要的生物大分子是 DNA、RNA 和蛋白質。蛋白質是細胞的“主力軍”,在酶中發揮著重要作用。DNA(脫氧核糖核酸)被認為是細胞的“藍圖”。它攜帶細胞生長、吸收營養物質和繁殖所需的所有遺傳資訊。RNA(核糖核酸)在細胞中充當“DNA 影印本”。
Biopython 提供 Bio.Sequence 物件,表示核苷酸,即 DNA 和 RNA 的構建塊。
Biopython - 安裝
本節說明如何在您的機器上安裝 Biopython。安裝非常簡單,不會超過五分鐘。
步驟 1 - 驗證 Python 安裝
Biopython 旨在與 Python 2.5 或更高版本一起使用。因此,必須首先安裝 python。在命令提示符中執行以下命令:
> python --version
定義如下:
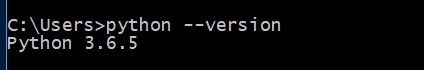
如果正確安裝,它將顯示 python 的版本。否則,下載最新版本的 python,安裝它,然後再次執行該命令。
步驟 2 - 使用 pip 安裝 Biopython
在所有平臺上,使用命令列透過 pip 安裝 Biopython 很容易。鍵入以下命令:
> pip install biopython
您將在螢幕上看到以下響應:
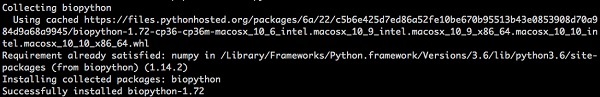
更新舊版本的 Biopython:
> pip install biopython –-upgrade
您將在螢幕上看到以下響應:
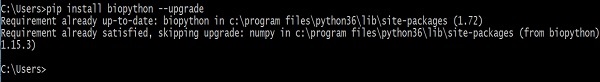
執行此命令後,將在安裝最新版本之前刪除舊版本的 Biopython 和 NumPy(Biopython 依賴於它)。
步驟 3 - 驗證 Biopython 安裝
現在,您已成功在您的機器上安裝了 Biopython。要驗證 Biopython 是否已正確安裝,請在 Python 控制檯中鍵入以下命令:
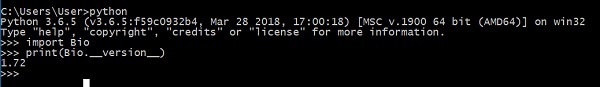
它顯示 Biopython 的版本。
備用方法 - 使用原始碼安裝 Biopython
要使用原始碼安裝 Biopython,請按照以下說明操作:
從以下連結下載 Biopython 的最新版本:https://biopython.org/wiki/Download
截至目前,最新版本為biopython-1.72。
下載檔案並解壓縮壓縮的存檔檔案,移動到原始碼資料夾並鍵入以下命令:
> python setup.py build
這將根據以下說明從原始碼構建 Biopython:
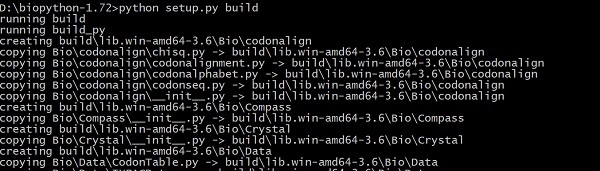
現在,使用以下命令測試程式碼:
> python setup.py test
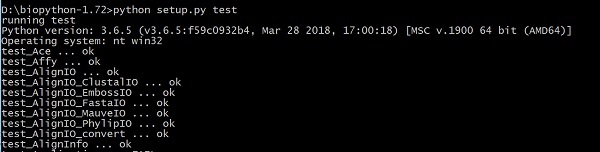
最後,使用以下命令進行安裝:
> python setup.py install
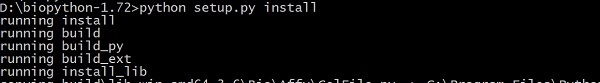
Biopython - 建立簡單的應用程式
讓我們建立一個簡單的 Biopython 應用程式來解析生物資訊學檔案並列印內容。這將幫助我們理解 Biopython 的一般概念以及它如何在生物資訊學領域提供幫助。
步驟 1 - 首先,建立一個樣本序列檔案“example.fasta”並將以下內容放入其中。
>sp|P25730|FMS1_ECOLI CS1 fimbrial subunit A precursor (CS1 pilin) MKLKKTIGAMALATLFATMGASAVEKTISVTASVDPTVDLLQSDGSALPNSVALTYSPAV NNFEAHTINTVVHTNDSDKGVVVKLSADPVLSNVLNPTLQIPVSVNFAGKPLSTTGITID SNDLNFASSGVNKVSSTQKLSIHADATRVTGGALTAGQYQGLVSIILTKSTTTTTTTKGT >sp|P15488|FMS3_ECOLI CS3 fimbrial subunit A precursor (CS3 pilin) MLKIKYLLIGLSLSAMSSYSLAAAGPTLTKELALNVLSPAALDATWAPQDNLTLSNTGVS NTLVGVLTLSNTSIDTVSIASTNVSDTSKNGTVTFAHETNNSASFATTISTDNANITLDK NAGNTIVKTTNGSQLPTNLPLKFITTEGNEHLVSGNYRANITITSTIKGGGTKKGTTDKK
副檔名fasta指的是序列檔案的格式。FASTA 源自生物資訊學軟體 FASTA,因此得名。FASTA 格式有多個序列一個接一個地排列,每個序列都有自己的 ID、名稱、描述和實際序列資料。
步驟 2 - 建立一個新的 Python 指令碼“simple_example.py”,輸入以下程式碼並儲存。
from Bio.SeqIO import parse
from Bio.SeqRecord import SeqRecord
from Bio.Seq import Seq
file = open("example.fasta")
records = parse(file, "fasta") for record in records:
print("Id: %s" % record.id)
print("Name: %s" % record.name)
print("Description: %s" % record.description)
print("Annotations: %s" % record.annotations)
print("Sequence Data: %s" % record.seq)
print("Sequence Alphabet: %s" % record.seq.alphabet)
讓我們更深入地瞭解一下程式碼:
第 1 行匯入 Bio.SeqIO 模組中可用的 parse 類。Bio.SeqIO 模組用於以不同的格式讀取和寫入序列檔案,而 `parse` 類用於解析序列檔案的內容。
第 2 行匯入 Bio.SeqRecord 模組中可用的 SeqRecord 類。此模組用於操作序列記錄,而 SeqRecord 類用於表示序列檔案中可用的特定序列。
“第 3 行”匯入 Bio.Seq 模組中可用的 Seq 類。此模組用於操作序列資料,而 Seq 類用於表示序列檔案中可用的特定序列記錄的序列資料。
第 5 行使用常規 Python 函式 open 開啟“example.fasta”檔案。
第 7 行解析序列檔案的內容,並將內容作為 SeqRecord 物件列表返回。
第 9-15 行使用 Python for 迴圈遍歷記錄,並列印序列記錄(SqlRecord)的屬性,例如 ID、名稱、描述、序列資料等。
第 15 行使用 Alphabet 類列印序列的型別。
步驟 3 - 開啟命令提示符並轉到包含序列檔案“example.fasta”的資料夾,然後執行以下命令:
> python simple_example.py
步驟 4 - Python 執行指令碼並列印樣本檔案“example.fasta”中可用的所有序列資料。輸出將類似於以下內容。
Id: sp|P25730|FMS1_ECOLI
Name: sp|P25730|FMS1_ECOLI
Decription: sp|P25730|FMS1_ECOLI CS1 fimbrial subunit A precursor (CS1 pilin)
Annotations: {}
Sequence Data: MKLKKTIGAMALATLFATMGASAVEKTISVTASVDPTVDLLQSDGSALPNSVALTYSPAVNNFEAHTINTVVHTNDSD
KGVVVKLSADPVLSNVLNPTLQIPVSVNFAGKPLSTTGITIDSNDLNFASSGVNKVSSTQKLSIHADATRVTGGALTA
GQYQGLVSIILTKSTTTTTTTKGT
Sequence Alphabet: SingleLetterAlphabet()
Id: sp|P15488|FMS3_ECOLI
Name: sp|P15488|FMS3_ECOLI
Decription: sp|P15488|FMS3_ECOLI CS3 fimbrial subunit A precursor (CS3 pilin)
Annotations: {}
Sequence Data: MLKIKYLLIGLSLSAMSSYSLAAAGPTLTKELALNVLSPAALDATWAPQDNLTLSNTGVSNTLVGVLTLSNTSIDTVS
IASTNVSDTSKNGTVTFAHETNNSASFATTISTDNANITLDKNAGNTIVKTTNGSQLPTNLPLKFITTEGNEHLVSGN
YRANITITSTIKGGGTKKGTTDKK
Sequence Alphabet: SingleLetterAlphabet()
在這個例子中,我們看到了三個類,parse、SeqRecord 和 Seq。這三個類提供了大部分功能,我們將在接下來的章節中學習這些類。
Biopython - 序列
序列是一系列字母,用於表示生物體的蛋白質、DNA 或 RNA。它由 Seq 類表示。Seq 類定義在 Bio.Seq 模組中。
讓我們在 Biopython 中建立一個簡單的序列,如下所示:
>>> from Bio.Seq import Seq
>>> seq = Seq("AGCT")
>>> seq
Seq('AGCT')
>>> print(seq)
AGCT
在這裡,我們建立了一個簡單的蛋白質序列AGCT,每個字母分別代表A賴氨酸、G甘氨酸、C半胱氨酸和T蘇氨酸。
每個 Seq 物件有兩個重要的屬性:
data - 實際序列字串 (AGCT)
alphabet - 用於表示序列型別。例如 DNA 序列、RNA 序列等。預設情況下,它不表示任何序列,並且本質上是通用的。
Alphabet 模組
Seq 物件包含 Alphabet 屬性以指定序列型別、字母和可能的運算。它定義在 Bio.Alphabet 模組中。Alphabet 可以定義如下:
>>> from Bio.Seq import Seq
>>> myseq = Seq("AGCT")
>>> myseq
Seq('AGCT')
>>> myseq.alphabet
Alphabet()
Alphabet 模組提供以下類來表示不同型別的序列。Alphabet - 所有型別字母的基類。
SingleLetterAlphabet - 長度為 1 的字母的通用字母表。它派生自 Alphabet,所有其他字母型別都派生自它。
>>> from Bio.Seq import Seq
>>> from Bio.Alphabet import single_letter_alphabet
>>> test_seq = Seq('AGTACACTGGT', single_letter_alphabet)
>>> test_seq
Seq('AGTACACTGGT', SingleLetterAlphabet())
ProteinAlphabet - 通用單字母蛋白質字母表。
>>> from Bio.Seq import Seq
>>> from Bio.Alphabet import generic_protein
>>> test_seq = Seq('AGTACACTGGT', generic_protein)
>>> test_seq
Seq('AGTACACTGGT', ProteinAlphabet())
NucleotideAlphabet - 通用單字母核苷酸字母表。
>>> from Bio.Seq import Seq
>>> from Bio.Alphabet import generic_nucleotide
>>> test_seq = Seq('AGTACACTGGT', generic_nucleotide) >>> test_seq
Seq('AGTACACTGGT', NucleotideAlphabet())
DNAAlphabet - 通用單字母 DNA 字母表。
>>> from Bio.Seq import Seq
>>> from Bio.Alphabet import generic_dna
>>> test_seq = Seq('AGTACACTGGT', generic_dna)
>>> test_seq
Seq('AGTACACTGGT', DNAAlphabet())
RNAAlphabet - 通用單字母 RNA 字母表。
>>> from Bio.Seq import Seq
>>> from Bio.Alphabet import generic_rna
>>> test_seq = Seq('AGTACACTGGT', generic_rna)
>>> test_seq
Seq('AGTACACTGGT', RNAAlphabet())
Biopython 模組 Bio.Alphabet.IUPAC 提供了由 IUPAC 社群定義的基本序列型別。它包含以下類:
IUPACProtein (protein) - 20 種標準氨基酸的 IUPAC 蛋白質字母表。
ExtendedIUPACProtein (extended_protein) - 包括 X 的擴充套件大寫 IUPAC 蛋白質單字母字母表。
IUPACAmbiguousDNA (ambiguous_dna) - 大寫 IUPAC 模糊 DNA。
IUPACUnambiguousDNA (unambiguous_dna) - 大寫 IUPAC 明確 DNA (GATC)。
ExtendedIUPACDNA (extended_dna) - 擴充套件 IUPAC DNA 字母表。
IUPACAmbiguousRNA (ambiguous_rna) − IUPAC模糊RNA大寫字母。
IUPACUnambiguousRNA (unambiguous_rna) − IUPAC明確RNA大寫字母 (GAUC)。
考慮以下所示的IUPACProtein類的簡單示例:
>>> from Bio.Alphabet import IUPAC
>>> protein_seq = Seq("AGCT", IUPAC.protein)
>>> protein_seq
Seq('AGCT', IUPACProtein())
>>> protein_seq.alphabet
此外,Biopython透過Bio.Data模組公開了所有與生物資訊學相關的配置資料。例如,IUPACData.protein_letters包含IUPACProtein字母表中可能的字母。
>>> from Bio.Data import IUPACData >>> IUPACData.protein_letters 'ACDEFGHIKLMNPQRSTVWY'
基本操作
本節簡要介紹了Seq類中可用的所有基本操作。序列類似於Python字串。我們可以在序列中執行Python字串操作,如切片、計數、連線、查詢、分割和去除首尾空格。
使用以下程式碼獲取各種輸出。
獲取序列中的第一個值。
>>> seq_string = Seq("AGCTAGCT")
>>> seq_string[0]
'A'
列印前兩個值。
>>> seq_string[0:2]
Seq('AG')
列印所有值。
>>> seq_string[ : ]
Seq('AGCTAGCT')
執行長度和計數操作。
>>> len(seq_string)
8
>>> seq_string.count('A')
2
新增兩個序列。
>>> from Bio.Alphabet import generic_dna, generic_protein
>>> seq1 = Seq("AGCT", generic_dna)
>>> seq2 = Seq("TCGA", generic_dna)
>>> seq1+seq2
Seq('AGCTTCGA', DNAAlphabet())
這裡,上面兩個序列物件seq1、seq2是通用DNA序列,因此您可以將它們新增並生成新的序列。您不能新增具有不相容字母的序列,例如如下所示的蛋白質序列和DNA序列:
>>> dna_seq = Seq('AGTACACTGGT', generic_dna)
>>> protein_seq = Seq('AGUACACUGGU', generic_protein)
>>> dna_seq + protein_seq
.....
.....
TypeError: Incompatible alphabets DNAAlphabet() and ProteinAlphabet()
>>>
要新增兩個或多個序列,首先將其儲存在Python列表中,然後使用“for迴圈”檢索它,最後將其加在一起,如下所示:
>>> from Bio.Alphabet import generic_dna
>>> list = [Seq("AGCT",generic_dna),Seq("TCGA",generic_dna),Seq("AAA",generic_dna)]
>>> for s in list:
... print(s)
...
AGCT
TCGA
AAA
>>> final_seq = Seq(" ",generic_dna)
>>> for s in list:
... final_seq = final_seq + s
...
>>> final_seq
Seq('AGCTTCGAAAA', DNAAlphabet())
在以下部分,提供了各種程式碼以根據需求獲取輸出。
更改序列的大小寫。
>>> from Bio.Alphabet import generic_rna
>>> rna = Seq("agct", generic_rna)
>>> rna.upper()
Seq('AGCT', RNAAlphabet())
檢查Python成員資格和身份運算子。
>>> rna = Seq("agct", generic_rna)
>>> 'a' in rna
True
>>> 'A' in rna
False
>>> rna1 = Seq("AGCT", generic_dna)
>>> rna is rna1
False
在給定序列中查詢單個字母或字母序列。
>>> protein_seq = Seq('AGUACACUGGU', generic_protein)
>>> protein_seq.find('G')
1
>>> protein_seq.find('GG')
8
執行分割操作。
>>> protein_seq = Seq('AGUACACUGGU', generic_protein)
>>> protein_seq.split('A')
[Seq('', ProteinAlphabet()), Seq('GU', ProteinAlphabet()),
Seq('C', ProteinAlphabet()), Seq('CUGGU', ProteinAlphabet())]
在序列中執行去除首尾空格操作。
>>> strip_seq = Seq(" AGCT ")
>>> strip_seq
Seq(' AGCT ')
>>> strip_seq.strip()
Seq('AGCT')
Biopython - 高階序列操作
在本章中,我們將討論Biopython提供的一些高階序列功能。
互補序列和反向互補序列
核苷酸序列可以反向互補以獲得新的序列。此外,互補序列可以反向互補以獲得原始序列。Biopython提供了兩種方法來實現此功能:complement和reverse_complement。程式碼如下所示:
>>> from Bio.Alphabet import IUPAC
>>> nucleotide = Seq('TCGAAGTCAGTC', IUPAC.ambiguous_dna)
>>> nucleotide.complement()
Seq('AGCTTCAGTCAG', IUPACAmbiguousDNA())
>>>
這裡,complement()方法允許互補DNA或RNA序列。reverse_complement()方法對從左到右的結果序列進行互補和反轉。如下所示:
>>> nucleotide.reverse_complement()
Seq('GACTGACTTCGA', IUPACAmbiguousDNA())
Biopython使用Bio.Data.IUPACData提供的ambiguous_dna_complement變數來執行互補操作。
>>> from Bio.Data import IUPACData
>>> import pprint
>>> pprint.pprint(IUPACData.ambiguous_dna_complement) {
'A': 'T',
'B': 'V',
'C': 'G',
'D': 'H',
'G': 'C',
'H': 'D',
'K': 'M',
'M': 'K',
'N': 'N',
'R': 'Y',
'S': 'S',
'T': 'A',
'V': 'B',
'W': 'W',
'X': 'X',
'Y': 'R'}
>>>
GC含量
基因組DNA鹼基組成(GC含量)預計會顯著影響基因組功能和物種生態。GC含量是GC核苷酸數量除以總核苷酸數量。
要獲取GC核苷酸含量,請匯入以下模組並執行以下步驟:
>>> from Bio.SeqUtils import GC
>>> nucleotide = Seq("GACTGACTTCGA",IUPAC.unambiguous_dna)
>>> GC(nucleotide)
50.0
轉錄
轉錄是將DNA序列轉換為RNA序列的過程。實際的生物轉錄過程是執行反向互補(TCAG → CUGA)以獲得mRNA,將DNA視為模板鏈。但是,在生物資訊學以及在Biopython中,我們通常直接使用編碼鏈,並且可以透過將字母T更改為U來獲取mRNA序列。
以上的一個簡單示例如下所示:
>>> from Bio.Seq import Seq
>>> from Bio.Seq import transcribe
>>> from Bio.Alphabet import IUPAC
>>> dna_seq = Seq("ATGCCGATCGTAT",IUPAC.unambiguous_dna) >>> transcribe(dna_seq)
Seq('AUGCCGAUCGUAU', IUPACUnambiguousRNA())
>>>
要反轉轉錄,將T更改為U,如下面的程式碼所示:
>>> rna_seq = transcribe(dna_seq)
>>> rna_seq.back_transcribe()
Seq('ATGCCGATCGTAT', IUPACUnambiguousDNA())
要獲取DNA模板鏈,請反向互補反轉錄的RNA,如下所示:
>>> rna_seq.back_transcribe().reverse_complement()
Seq('ATACGATCGGCAT', IUPACUnambiguousDNA())
翻譯
翻譯是將RNA序列翻譯成蛋白質序列的過程。考慮以下所示的RNA序列:
>>> rna_seq = Seq("AUGGCCAUUGUAAU",IUPAC.unambiguous_rna)
>>> rna_seq
Seq('AUGGCCAUUGUAAUGGGCCGCUGAAAGGGUGCCCGAUAG', IUPACUnambiguousRNA())
現在,將translate()函式應用於上面的程式碼:
>>> rna_seq.translate()
Seq('MAIV', IUPACProtein())
上述RNA序列很簡單。考慮RNA序列AUGGCCAUUGUAAUGGGCCGCUGAAAGGGUGCCCGA並應用translate():
>>> rna = Seq('AUGGCCAUUGUAAUGGGCCGCUGAAAGGGUGCCCGA', IUPAC.unambiguous_rna)
>>> rna.translate()
Seq('MAIVMGR*KGAR', HasStopCodon(IUPACProtein(), '*'))
這裡,終止密碼子用星號“*”表示。
在translate()方法中,可以停止在第一個終止密碼子處。要執行此操作,您可以在translate()中分配to_stop=True,如下所示:
>>> rna.translate(to_stop = True)
Seq('MAIVMGR', IUPACProtein())
這裡,終止密碼子不包含在結果序列中,因為它不包含一個。
翻譯表
NCBI的遺傳密碼頁面提供了Biopython使用的翻譯表的完整列表。讓我們看一個標準表的示例以視覺化程式碼:
>>> from Bio.Data import CodonTable >>> table = CodonTable.unambiguous_dna_by_name["Standard"] >>> print(table) Table 1 Standard, SGC0 | T | C | A | G | --+---------+---------+---------+---------+-- T | TTT F | TCT S | TAT Y | TGT C | T T | TTC F | TCC S | TAC Y | TGC C | C T | TTA L | TCA S | TAA Stop| TGA Stop| A T | TTG L(s)| TCG S | TAG Stop| TGG W | G --+---------+---------+---------+---------+-- C | CTT L | CCT P | CAT H | CGT R | T C | CTC L | CCC P | CAC H | CGC R | C C | CTA L | CCA P | CAA Q | CGA R | A C | CTG L(s)| CCG P | CAG Q | CGG R | G --+---------+---------+---------+---------+-- A | ATT I | ACT T | AAT N | AGT S | T A | ATC I | ACC T | AAC N | AGC S | C A | ATA I | ACA T | AAA K | AGA R | A A | ATG M(s)| ACG T | AAG K | AGG R | G --+---------+---------+---------+---------+-- G | GTT V | GCT A | GAT D | GGT G | T G | GTC V | GCC A | GAC D | GGC G | C G | GTA V | GCA A | GAA E | GGA G | A G | GTG V | GCG A | GAG E | GGG G | G --+---------+---------+---------+---------+-- >>>
Biopython使用此表將DNA翻譯成蛋白質以及查詢終止密碼子。
Biopython - 序列I/O操作
Biopython提供了一個模組Bio.SeqIO,分別用於從檔案(任何流)讀取和寫入序列。它支援生物資訊學中幾乎所有可用的檔案格式。大多數軟體為不同的檔案格式提供了不同的方法。但是,Biopython有意識地遵循單一方法,透過其SeqRecord物件向用戶呈現解析的序列資料。
讓我們在下一節中進一步瞭解SeqRecord。
SeqRecord
Bio.SeqRecord模組提供SeqRecord來儲存序列的元資訊以及序列資料本身,如下所示:
seq − 它是實際的序列。
id − 它是給定序列的主要識別符號。預設型別為字串。
name − 它是序列的名稱。預設型別為字串。
description − 它顯示有關序列的人類可讀資訊。
annotations − 它是有關序列的其他資訊的字典。
SeqRecord可以按以下方式匯入
from Bio.SeqRecord import SeqRecord
讓我們在接下來的部分中瞭解使用真實序列檔案解析序列檔案的細微差別。
解析序列檔案格式
本節說明如何解析兩種最流行的序列檔案格式:FASTA和GenBank。
FASTA
FASTA是用於儲存序列資料的最基本的檔案格式。最初,FASTA是一個用於DNA和蛋白質序列比對的軟體包,開發於生物資訊學的早期發展階段,主要用於搜尋序列相似性。
Biopython提供了一個示例FASTA檔案,可以在以下位置訪問:https://github.com/biopython/biopython/blob/master/Doc/examples/ls_orchid.fasta.
將此檔案下載並儲存到您的Biopython示例目錄中,命名為“orchid.fasta”。
Bio.SeqIO模組提供parse()方法來處理序列檔案,可以按如下方式匯入:
from Bio.SeqIO import parse
parse()方法包含兩個引數,第一個是檔案控制代碼,第二個是檔案格式。
>>> file = open('path/to/biopython/sample/orchid.fasta')
>>> for record in parse(file, "fasta"):
... print(record.id)
...
gi|2765658|emb|Z78533.1|CIZ78533
gi|2765657|emb|Z78532.1|CCZ78532
..........
..........
gi|2765565|emb|Z78440.1|PPZ78440
gi|2765564|emb|Z78439.1|PBZ78439
>>>
這裡,parse()方法返回一個可迭代物件,該物件在每次迭代時返回SeqRecord。作為可迭代物件,它提供了許多複雜且易用的方法,讓我們看看其中的一些功能。
next()
next()方法返回可迭代物件中可用的下一個專案,我們可以使用它來獲取第一個序列,如下所示:
>>> first_seq_record = next(SeqIO.parse(open('path/to/biopython/sample/orchid.fasta'),'fasta'))
>>> first_seq_record.id 'gi|2765658|emb|Z78533.1|CIZ78533'
>>> first_seq_record.name 'gi|2765658|emb|Z78533.1|CIZ78533'
>>> first_seq_record.seq Seq('CGTAACAAGGTTTCCGTAGGTGAACCTGCGGAAGGATCATTGATGAGACCGTGG...CGC', SingleLetterAlphabet())
>>> first_seq_record.description 'gi|2765658|emb|Z78533.1|CIZ78533 C.irapeanum 5.8S rRNA gene and ITS1 and ITS2 DNA'
>>> first_seq_record.annotations
{}
>>>
這裡,seq_record.annotations為空,因為FASTA格式不支援序列註釋。
列表推導式
我們可以使用列表推導式將可迭代物件轉換為列表,如下所示
>>> seq_iter = SeqIO.parse(open('path/to/biopython/sample/orchid.fasta'),'fasta')
>>> all_seq = [seq_record for seq_record in seq_iter] >>> len(all_seq)
94
>>>
這裡,我們使用了len方法來獲取總數。我們可以獲取長度最大的序列,如下所示:
>>> seq_iter = SeqIO.parse(open('path/to/biopython/sample/orchid.fasta'),'fasta')
>>> max_seq = max(len(seq_record.seq) for seq_record in seq_iter)
>>> max_seq
789
>>>
我們也可以使用以下程式碼過濾序列:
>>> seq_iter = SeqIO.parse(open('path/to/biopython/sample/orchid.fasta'),'fasta')
>>> seq_under_600 = [seq_record for seq_record in seq_iter if len(seq_record.seq) < 600]
>>> for seq in seq_under_600:
... print(seq.id)
...
gi|2765606|emb|Z78481.1|PIZ78481
gi|2765605|emb|Z78480.1|PGZ78480
gi|2765601|emb|Z78476.1|PGZ78476
gi|2765595|emb|Z78470.1|PPZ78470
gi|2765594|emb|Z78469.1|PHZ78469
gi|2765564|emb|Z78439.1|PBZ78439
>>>
將SqlRecord物件(解析資料)的集合寫入檔案就像呼叫SeqIO.write方法一樣簡單,如下所示:
file = open("converted.fasta", "w)
SeqIO.write(seq_record, file, "fasta")
此方法可以有效地用於轉換格式,如下所示:
file = open("converted.gbk", "w)
SeqIO.write(seq_record, file, "genbank")
GenBank
它是一種更豐富的基因序列格式,包括用於各種註釋的欄位。Biopython提供了一個示例GenBank檔案,可以在以下位置訪問:https://github.com/biopython/biopython/blob/master/Doc/examples/ls_orchid.fasta.
將檔案下載並儲存到您的Biopython示例目錄中,命名為“orchid.gbk”
由於Biopython提供了一個單一函式parse來解析所有生物資訊學格式,因此解析GenBank格式就像在parse方法中更改格式選項一樣簡單。
相同的程式碼如下所示:
>>> from Bio import SeqIO
>>> from Bio.SeqIO import parse
>>> seq_record = next(parse(open('path/to/biopython/sample/orchid.gbk'),'genbank'))
>>> seq_record.id
'Z78533.1'
>>> seq_record.name
'Z78533'
>>> seq_record.seq Seq('CGTAACAAGGTTTCCGTAGGTGAACCTGCGGAAGGATCATTGATGAGACCGTGG...CGC', IUPACAmbiguousDNA())
>>> seq_record.description
'C.irapeanum 5.8S rRNA gene and ITS1 and ITS2 DNA'
>>> seq_record.annotations {
'molecule_type': 'DNA',
'topology': 'linear',
'data_file_division': 'PLN',
'date': '30-NOV-2006',
'accessions': ['Z78533'],
'sequence_version': 1,
'gi': '2765658',
'keywords': ['5.8S ribosomal RNA', '5.8S rRNA gene', 'internal transcribed spacer', 'ITS1', 'ITS2'],
'source': 'Cypripedium irapeanum',
'organism': 'Cypripedium irapeanum',
'taxonomy': [
'Eukaryota',
'Viridiplantae',
'Streptophyta',
'Embryophyta',
'Tracheophyta',
'Spermatophyta',
'Magnoliophyta',
'Liliopsida',
'Asparagales',
'Orchidaceae',
'Cypripedioideae',
'Cypripedium'],
'references': [
Reference(title = 'Phylogenetics of the slipper orchids (Cypripedioideae:
Orchidaceae): nuclear rDNA ITS sequences', ...),
Reference(title = 'Direct Submission', ...)
]
}
Biopython - 序列比對
序列比對是將兩個或多個序列(DNA、RNA或蛋白質序列)按特定順序排列以識別它們之間相似區域的過程。
識別相似區域使我們能夠推斷出許多資訊,例如不同物種之間哪些性狀是保守的、不同物種在遺傳上有多接近、物種如何進化等等。Biopython為序列比對提供了廣泛的支援。
讓我們在本節中學習Biopython提供的一些重要功能:
解析序列比對
Biopython提供了一個模組Bio.AlignIO來讀取和寫入序列比對。在生物資訊學中,有很多格式可用於指定序列比對資料,類似於前面學習的序列資料。Bio.AlignIO提供類似於Bio.SeqIO的API,不同之處在於Bio.SeqIO處理序列資料,而Bio.AlignIO處理序列比對資料。
在開始學習之前,讓我們從網際網路下載一個示例序列比對檔案。
要下載示例檔案,請按照以下步驟操作:
步驟1 − 開啟您喜歡的瀏覽器並訪問http://pfam.xfam.org/family/browse網站。它將按字母順序顯示所有Pfam家族。
步驟2 − 選擇任何一個種子值較少的家族。它包含最少的資料,使我們能夠輕鬆地處理比對。這裡,我們選擇了/點選了PF18225,它會開啟http://pfam.xfam.org/family/PF18225並顯示有關它的完整詳細資訊,包括序列比對。
步驟3 − 轉到比對部分,並以Stockholm格式(PF18225_seed.txt)下載序列比對檔案。
讓我們嘗試使用Bio.AlignIO讀取下載的序列比對檔案,如下所示:
匯入Bio.AlignIO模組
>>> from Bio import AlignIO
使用read方法讀取比對。read方法用於讀取給定檔案中可用的單個比對資料。如果給定檔案包含多個比對,我們可以使用parse方法。parse方法返回可迭代的比對物件,類似於Bio.SeqIO模組中的parse方法。
>>> alignment = AlignIO.read(open("PF18225_seed.txt"), "stockholm")
列印比對物件。
>>> print(alignment) SingleLetterAlphabet() alignment with 6 rows and 65 columns MQNTPAERLPAIIEKAKSKHDINVWLLDRQGRDLLEQRVPAKVA...EGP B7RZ31_9GAMM/59-123 AKQRGIAGLEEWLHRLDHSEAIPIFLIDEAGKDLLEREVPADIT...KKP A0A0C3NPG9_9PROT/58-119 ARRHGQEYFQQWLERQPKKVKEQVFAVDQFGRELLGRPLPEDMA...KKP A0A143HL37_9GAMM/57-121 TRRHGPESFRFWLERQPVEARDRIYAIDRSGAEILDRPIPRGMA...NKP A0A0X3UC67_9GAMM/57-121 AINRNTQQLTQDLRAMPNWSLRFVYIVDRNNQDLLKRPLPPGIM...NRK B3PFT7_CELJU/62-126 AVNATEREFTERIRTLPHWARRNVFVLDSQGFEIFDRELPSPVA...NRT K4KEM7_SIMAS/61-125 >>>
我們還可以檢查比對中可用的序列(SeqRecord),如下所示:
>>> for align in alignment: ... print(align.seq) ... MQNTPAERLPAIIEKAKSKHDINVWLLDRQGRDLLEQRVPAKVATVANQLRGRKRRAFARHREGP AKQRGIAGLEEWLHRLDHSEAIPIFLIDEAGKDLLEREVPADITA---RLDRRREHGEHGVRKKP ARRHGQEYFQQWLERQPKKVKEQVFAVDQFGRELLGRPLPEDMAPMLIALNYRNRESHAQVDKKP TRRHGPESFRFWLERQPVEARDRIYAIDRSGAEILDRPIPRGMAPLFKVLSFRNREDQGLVNNKP AINRNTQQLTQDLRAMPNWSLRFVYIVDRNNQDLLKRPLPPGIMVLAPRLTAKHPYDKVQDRNRK AVNATEREFTERIRTLPHWARRNVFVLDSQGFEIFDRELPSPVADLMRKLDLDRPFKKLERKNRT >>>
多序列比對
通常,大多數序列比對檔案包含單個比對資料,使用read方法解析就足夠了。在多序列比對的概念中,兩個或多個序列被比較以找到它們之間最佳的子序列匹配,並最終將結果以多序列比對的形式儲存在一個檔案中。
如果輸入序列比對格式包含多個序列比對,那麼我們需要使用parse方法而不是read方法,如下所示:
>>> from Bio import AlignIO
>>> alignments = AlignIO.parse(open("PF18225_seed.txt"), "stockholm")
>>> print(alignments)
<generator object parse at 0x000001CD1C7E0360>
>>> for alignment in alignments:
... print(alignment)
...
SingleLetterAlphabet() alignment with 6 rows and 65 columns
MQNTPAERLPAIIEKAKSKHDINVWLLDRQGRDLLEQRVPAKVA...EGP B7RZ31_9GAMM/59-123
AKQRGIAGLEEWLHRLDHSEAIPIFLIDEAGKDLLEREVPADIT...KKP A0A0C3NPG9_9PROT/58-119
ARRHGQEYFQQWLERQPKKVKEQVFAVDQFGRELLGRPLPEDMA...KKP A0A143HL37_9GAMM/57-121
TRRHGPESFRFWLERQPVEARDRIYAIDRSGAEILDRPIPRGMA...NKP A0A0X3UC67_9GAMM/57-121
AINRNTQQLTQDLRAMPNWSLRFVYIVDRNNQDLLKRPLPPGIM...NRK B3PFT7_CELJU/62-126
AVNATEREFTERIRTLPHWARRNVFVLDSQGFEIFDRELPSPVA...NRT K4KEM7_SIMAS/61-125
>>>
在這裡,parse方法返回可迭代的比對物件,並且可以對其進行迭代以獲取實際的比對。
成對序列比對
成對序列比對一次只比較兩個序列,並提供最佳可能的序列比對。成對比對易於理解,並且從所得的序列比對結果中推斷出資訊也十分便捷。
Biopython提供了一個特殊的模組Bio.pairwise2,用於使用成對方法識別比對序列。Biopython應用最佳演算法來查詢比對序列,其效能與其他軟體相當。
讓我們編寫一個示例,使用成對模組查詢兩個簡單且假設的序列的序列比對。這將幫助我們理解序列比對的概念以及如何使用Biopython對其進行程式設計。
步驟 1
使用以下命令匯入模組pairwise2:
>>> from Bio import pairwise2
步驟 2
建立兩個序列,seq1和seq2:
>>> from Bio.Seq import Seq
>>> seq1 = Seq("ACCGGT")
>>> seq2 = Seq("ACGT")
步驟 3
使用以下程式碼行呼叫pairwise2.align.globalxx方法,並傳入seq1和seq2以查詢比對:
>>> alignments = pairwise2.align.globalxx(seq1, seq2)
在這裡,globalxx方法執行實際工作,並找到給定序列中所有可能的最佳比對。實際上,Bio.pairwise2提供了相當一組方法,這些方法遵循以下約定在不同情況下查詢比對。
<sequence alignment type>XY
這裡,序列比對型別指的是比對型別,可以是全域性或區域性。全域性型別透過考慮整個序列來查詢序列比對。區域性型別透過檢視給定序列的子集來查詢序列比對。這將比較繁瑣,但提供了關於給定序列之間相似性的更佳理解。
X指的是匹配得分。可能的值有x(完全匹配)、m(基於相同字元的得分)、d(使用者提供的包含字元和匹配得分的字典)以及c(使用者自定義函式,用於提供自定義評分演算法)。
Y指的是間隔罰分。可能的值有x(無間隔罰分)、s(兩個序列的罰分相同)、d(每個序列的罰分不同)以及c(使用者自定義函式,用於提供自定義間隔罰分)。
因此,localds也是一個有效的方法,它使用區域性比對技術、使用者提供的匹配字典和使用者提供的兩個序列的間隔罰分來查詢序列比對。
>>> test_alignments = pairwise2.align.localds(seq1, seq2, blosum62, -10, -1)
這裡,blosum62指的是pairwise2模組中提供匹配得分的字典。-10指的是間隔開啟罰分,-1指的是間隔擴充套件罰分。
步驟 4
遍歷可迭代的比對物件,獲取每個單獨的比對物件並列印它。
>>> for alignment in alignments:
... print(alignment)
...
('ACCGGT', 'A-C-GT', 4.0, 0, 6)
('ACCGGT', 'AC--GT', 4.0, 0, 6)
('ACCGGT', 'A-CG-T', 4.0, 0, 6)
('ACCGGT', 'AC-G-T', 4.0, 0, 6)
步驟 5
Bio.pairwise2模組提供了一個格式化方法format_alignment,可以更好地視覺化結果:
>>> from Bio.pairwise2 import format_alignment >>> alignments = pairwise2.align.globalxx(seq1, seq2) >>> for alignment in alignments: ... print(format_alignment(*alignment)) ... ACCGGT | | || A-C-GT Score=4 ACCGGT || || AC--GT Score=4 ACCGGT | || | A-CG-T Score=4 ACCGGT || | | AC-G-T Score=4 >>>
Biopython還提供另一個模組來進行序列比對,即Align。此模組提供了一組不同的API來簡化引數的設定,例如演算法、模式、匹配得分、間隔罰分等。對Align物件的簡單瞭解如下:
>>> from Bio import Align >>> aligner = Align.PairwiseAligner() >>> print(aligner) Pairwise sequence aligner with parameters match score: 1.000000 mismatch score: 0.000000 target open gap score: 0.000000 target extend gap score: 0.000000 target left open gap score: 0.000000 target left extend gap score: 0.000000 target right open gap score: 0.000000 target right extend gap score: 0.000000 query open gap score: 0.000000 query extend gap score: 0.000000 query left open gap score: 0.000000 query left extend gap score: 0.000000 query right open gap score: 0.000000 query right extend gap score: 0.000000 mode: global >>>
對序列比對工具的支援
Biopython透過Bio.Align.Applications模組為許多序列比對工具提供了介面。一些工具列在下面:
- ClustalW
- MUSCLE
- EMBOSS needle和water
讓我們在Biopython中編寫一個簡單的示例,透過最流行的比對工具ClustalW建立序列比對。
步驟 1 - 從http://www.clustal.org/download/current/下載Clustalw程式並安裝它。此外,使用“clustal”安裝路徑更新系統PATH。
步驟 2 - 從模組Bio.Align.Applications匯入ClustalwCommanLine。
>>> from Bio.Align.Applications import ClustalwCommandline
步驟 3 - 透過呼叫ClustalwCommanLine並傳入輸入檔案opuntia.fasta(位於Biopython包中)來設定cmd。https://raw.githubusercontent.com/biopython/biopython/master/Doc/examples/opuntia.fasta
>>> cmd = ClustalwCommandline("clustalw2",
infile="/path/to/biopython/sample/opuntia.fasta")
>>> print(cmd)
clustalw2 -infile=fasta/opuntia.fasta
步驟 4 - 呼叫cmd()將執行clustalw命令,並輸出結果比對檔案opuntia.aln。
>>> stdout, stderr = cmd()
步驟 5 - 讀取並列印比對檔案,如下所示:
>>> from Bio import AlignIO
>>> align = AlignIO.read("/path/to/biopython/sample/opuntia.aln", "clustal")
>>> print(align)
SingleLetterAlphabet() alignment with 7 rows and 906 columns
TATACATTAAAGAAGGGGGATGCGGATAAATGGAAAGGCGAAAG...AGA
gi|6273285|gb|AF191659.1|AF191
TATACATTAAAGAAGGGGGATGCGGATAAATGGAAAGGCGAAAG...AGA
gi|6273284|gb|AF191658.1|AF191
TATACATTAAAGAAGGGGGATGCGGATAAATGGAAAGGCGAAAG...AGA
gi|6273287|gb|AF191661.1|AF191
TATACATAAAAGAAGGGGGATGCGGATAAATGGAAAGGCGAAAG...AGA
gi|6273286|gb|AF191660.1|AF191
TATACATTAAAGGAGGGGGATGCGGATAAATGGAAAGGCGAAAG...AGA
gi|6273290|gb|AF191664.1|AF191
TATACATTAAAGGAGGGGGATGCGGATAAATGGAAAGGCGAAAG...AGA
gi|6273289|gb|AF191663.1|AF191
TATACATTAAAGGAGGGGGATGCGGATAAATGGAAAGGCGAAAG...AGA
gi|6273291|gb|AF191665.1|AF191
>>>
Biopython - BLAST 概述
BLAST代表Basic Local Alignment Search Tool(基本區域性比對搜尋工具)。它查詢生物序列之間相似性區域。Biopython提供Bio.Blast模組來處理NCBI BLAST操作。您可以在本地連線或透過Internet連線執行BLAST。
讓我們在下一節中簡要了解這兩種連線:
透過Internet執行
Biopython提供Bio.Blast.NCBIWWW模組來呼叫BLAST的線上版本。為此,我們需要匯入以下模組:
>>> from Bio.Blast import NCBIWWW
NCBIWW模組提供qblast函式來查詢BLAST線上版本,https://blast.ncbi.nlm.nih.gov/Blast.cgi。qblast支援線上版本支援的所有引數。
要獲取有關此模組的任何幫助,請使用以下命令並瞭解其功能:
>>> help(NCBIWWW.qblast) Help on function qblast in module Bio.Blast.NCBIWWW: qblast( program, database, sequence, url_base = 'https://blast.ncbi.nlm.nih.gov/Blast.cgi', auto_format = None, composition_based_statistics = None, db_genetic_code = None, endpoints = None, entrez_query = '(none)', expect = 10.0, filter = None, gapcosts = None, genetic_code = None, hitlist_size = 50, i_thresh = None, layout = None, lcase_mask = None, matrix_name = None, nucl_penalty = None, nucl_reward = None, other_advanced = None, perc_ident = None, phi_pattern = None, query_file = None, query_believe_defline = None, query_from = None, query_to = None, searchsp_eff = None, service = None, threshold = None, ungapped_alignment = None, word_size = None, alignments = 500, alignment_view = None, descriptions = 500, entrez_links_new_window = None, expect_low = None, expect_high = None, format_entrez_query = None, format_object = None, format_type = 'XML', ncbi_gi = None, results_file = None, show_overview = None, megablast = None, template_type = None, template_length = None ) BLAST search using NCBI's QBLAST server or a cloud service provider. Supports all parameters of the qblast API for Put and Get. Please note that BLAST on the cloud supports the NCBI-BLAST Common URL API (http://ncbi.github.io/blast-cloud/dev/api.html). To use this feature, please set url_base to 'http://host.my.cloud.service.provider.com/cgi-bin/blast.cgi' and format_object = 'Alignment'. For more details, please see 8. Biopython – Overview of BLAST https://blast.ncbi.nlm.nih.gov/Blast.cgi?PAGE_TYPE = BlastDocs&DOC_TYPE = CloudBlast Some useful parameters: - program blastn, blastp, blastx, tblastn, or tblastx (lower case) - database Which database to search against (e.g. "nr"). - sequence The sequence to search. - ncbi_gi TRUE/FALSE whether to give 'gi' identifier. - descriptions Number of descriptions to show. Def 500. - alignments Number of alignments to show. Def 500. - expect An expect value cutoff. Def 10.0. - matrix_name Specify an alt. matrix (PAM30, PAM70, BLOSUM80, BLOSUM45). - filter "none" turns off filtering. Default no filtering - format_type "HTML", "Text", "ASN.1", or "XML". Def. "XML". - entrez_query Entrez query to limit Blast search - hitlist_size Number of hits to return. Default 50 - megablast TRUE/FALSE whether to use MEga BLAST algorithm (blastn only) - service plain, psi, phi, rpsblast, megablast (lower case) This function does no checking of the validity of the parameters and passes the values to the server as is. More help is available at: https://ncbi.github.io/blast-cloud/dev/api.html
通常,qblast函式的引數基本上類似於您可以在BLAST網頁上設定的不同引數。這使得qblast函式易於理解,並減少了使用它的學習曲線。
連線和搜尋
為了理解連線和搜尋BLAST線上版本的流程,讓我們透過Biopython對線上BLAST伺服器進行一個簡單的序列搜尋(在我們本地的序列檔案中可用)。
步驟 1 - 在Biopython目錄中建立一個名為blast_example.fasta的檔案,並將以下序列資訊作為輸入。
Example of a single sequence in FASTA/Pearson format: >sequence A ggtaagtcctctagtacaaacacccccaatattgtgatataattaaaattatattcatat tctgttgccagaaaaaacacttttaggctatattagagccatcttctttgaagcgttgtc >sequence B ggtaagtcctctagtacaaacacccccaatattgtgatataattaaaattatattca tattctgttgccagaaaaaacacttttaggctatattagagccatcttctttgaagcgttgtc
步驟 2 - 匯入NCBIWWW模組。
>>> from Bio.Blast import NCBIWWW
步驟 3 - 使用python IO模組開啟序列檔案blast_example.fasta。
>>> sequence_data = open("blast_example.fasta").read()
>>> sequence_data
'Example of a single sequence in FASTA/Pearson format:\n\n\n> sequence
A\nggtaagtcctctagtacaaacacccccaatattgtgatataattaaaatt
atattcatat\ntctgttgccagaaaaaacacttttaggctatattagagccatcttctttg aagcgttgtc\n\n'
步驟 4 - 現在,呼叫qblast函式,並將序列資料作為主要引數傳遞。其他引數表示資料庫(nt)和內部程式(blastn)。
>>> result_handle = NCBIWWW.qblast("blastn", "nt", sequence_data)
>>> result_handle
<_io.StringIO object at 0x000001EC9FAA4558>
blast_results儲存我們搜尋的結果。它可以儲存到檔案中以供以後使用,也可以解析以獲取詳細資訊。我們將在下一節中學習如何執行此操作。
步驟 5 - 同樣的功能也可以使用Seq物件來完成,而不是使用整個fasta檔案,如下所示:
>>> from Bio import SeqIO
>>> seq_record = next(SeqIO.parse(open('blast_example.fasta'),'fasta'))
>>> seq_record.id
'sequence'
>>> seq_record.seq
Seq('ggtaagtcctctagtacaaacacccccaatattgtgatataattaaaattatat...gtc',
SingleLetterAlphabet())
現在,呼叫qblast函式,並將Seq物件record.seq作為主要引數傳遞。
>>> result_handle = NCBIWWW.qblast("blastn", "nt", seq_record.seq)
>>> print(result_handle)
<_io.StringIO object at 0x000001EC9FAA4558>
BLAST將自動為您的序列分配一個識別符號。
步驟 6 - result_handle物件將包含整個結果,並可以儲存到檔案中以供以後使用。
>>> with open('results.xml', 'w') as save_file:
>>> blast_results = result_handle.read()
>>> save_file.write(blast_results)
我們將在後面的章節中瞭解如何解析結果檔案。
執行獨立的BLAST
本節介紹如何在本地系統中執行BLAST。如果您在本地系統中執行BLAST,它可能會更快,並且允許您建立自己的資料庫來搜尋序列。
連線BLAST
通常,由於BLAST體積龐大、執行軟體需要額外工作以及成本問題,不建議在本地執行。對於基本和高階用途,線上BLAST已足夠。當然,有時您可能需要在本地安裝它。
假設您正在進行頻繁的線上搜尋,這可能需要大量時間和網路流量,並且如果您有專有的序列資料或與IP相關的隱私問題,那麼建議在本地安裝它。
為此,我們需要遵循以下步驟:
步驟 1 - 使用給定的連結下載並安裝最新的blast二進位制檔案:ftp://ftp.ncbi.nlm.nih.gov/blast/executables/blast+/LATEST/
步驟 2 - 使用以下連結下載並解壓縮最新的必要資料庫:ftp://ftp.ncbi.nlm.nih.gov/blast/db/
BLAST軟體在其網站上提供了許多資料庫。讓我們從blast資料庫站點下載alu.n.gz檔案並將其解壓縮到alu資料夾中。此檔案為FASTA格式。要在我們的blast應用程式中使用此檔案,我們需要先將檔案從FASTA格式轉換為blast資料庫格式。BLAST提供makeblastdb應用程式來執行此轉換。
使用以下程式碼片段:
cd /path/to/alu makeblastdb -in alu.n -parse_seqids -dbtype nucl -out alun
執行以上程式碼將解析輸入檔案alu.n並建立BLAST資料庫,如多個檔案alun.nsq、alun.nsi等。現在,我們可以查詢此資料庫以查詢序列。
我們已在本地伺服器上安裝了BLAST,並且還擁有示例BLAST資料庫alun,可以對其進行查詢。
步驟 3 - 讓我們建立一個示例序列檔案來查詢資料庫。建立一個名為search.fsa的檔案,並將以下資料放入其中。
>gnl|alu|Z15030_HSAL001056 (Alu-J) AGGCTGGCACTGTGGCTCATGCTGAAATCCCAGCACGGCGGAGGACGGCGGAAGATTGCT TGAGCCTAGGAGTTTGCGACCAGCCTGGGTGACATAGGGAGATGCCTGTCTCTACGCAAA AGAAAAAAAAAATAGCTCTGCTGGTGGTGCATGCCTATAGTCTCAGCTATCAGGAGGCTG GGACAGGAGGATCACTTGGGCCCGGGAGTTGAGGCTGTGGTGAGCCACGATCACACCACT GCACTCCAGCCTGGGTGACAGAGCAAGACCCTGTCTCAAAACAAACAAATAA >gnl|alu|D00596_HSAL003180 (Alu-Sx) AGCCAGGTGTGGTGGCTCACGCCTGTAATCCCACCGCTTTGGGAGGCTGAGTCAGATCAC CTGAGGTTAGGAATTTGGGACCAGCCTGGCCAACATGGCGACACCCCAGTCTCTACTAAT AACACAAAAAATTAGCCAGGTGTGCTGGTGCATGTCTGTAATCCCAGCTACTCAGGAGGC TGAGGCATGAGAATTGCTCACGAGGCGGAGGTTGTAGTGAGCTGAGATCGTGGCACTGTA CTCCAGCCTGGCGACAGAGGGAGAACCCATGTCAAAAACAAAAAAAGACACCACCAAAGG TCAAAGCATA >gnl|alu|X55502_HSAL000745 (Alu-J) TGCCTTCCCCATCTGTAATTCTGGCACTTGGGGAGTCCAAGGCAGGATGATCACTTATGC CCAAGGAATTTGAGTACCAAGCCTGGGCAATATAACAAGGCCCTGTTTCTACAAAAACTT TAAACAATTAGCCAGGTGTGGTGGTGCGTGCCTGTGTCCAGCTACTCAGGAAGCTGAGGC AAGAGCTTGAGGCTACAGTGAGCTGTGTTCCACCATGGTGCTCCAGCCTGGGTGACAGGG CAAGACCCTGTCAAAAGAAAGGAAGAAAGAACGGAAGGAAAGAAGGAAAGAAACAAGGAG AG
序列資料是從alu.n檔案中收集的;因此,它與我們的資料庫匹配。
步驟 4 - BLAST軟體提供了許多應用程式來搜尋資料庫,我們使用blastn。blastn應用程式至少需要三個引數:db、query和out。db指的是要搜尋的資料庫;query是要匹配的序列;out是要儲存結果的檔案。現在,執行以下命令執行此簡單查詢:
blastn -db alun -query search.fsa -out results.xml -outfmt 5
執行以上命令將在results.xml檔案中搜索並輸出結果,如下所示(部分資料):
<?xml version = "1.0"?>
<!DOCTYPE BlastOutput PUBLIC "-//NCBI//NCBI BlastOutput/EN"
"http://www.ncbi.nlm.nih.gov/dtd/NCBI_BlastOutput.dtd">
<BlastOutput>
<BlastOutput_program>blastn</BlastOutput_program>
<BlastOutput_version>BLASTN 2.7.1+</BlastOutput_version>
<BlastOutput_reference>Zheng Zhang, Scott Schwartz, Lukas Wagner, and Webb
Miller (2000), "A greedy algorithm for aligning DNA sequences", J
Comput Biol 2000; 7(1-2):203-14.
</BlastOutput_reference>
<BlastOutput_db>alun</BlastOutput_db>
<BlastOutput_query-ID>Query_1</BlastOutput_query-ID>
<BlastOutput_query-def>gnl|alu|Z15030_HSAL001056 (Alu-J)</BlastOutput_query-def>
<BlastOutput_query-len>292</BlastOutput_query-len>
<BlastOutput_param>
<Parameters>
<Parameters_expect>10</Parameters_expect>
<Parameters_sc-match>1</Parameters_sc-match>
<Parameters_sc-mismatch>-2</Parameters_sc-mismatch>
<Parameters_gap-open>0</Parameters_gap-open>
<Parameters_gap-extend>0</Parameters_gap-extend>
<Parameters_filter>L;m;</Parameters_filter>
</Parameters>
</BlastOutput_param>
<BlastOutput_iterations>
<Iteration>
<Iteration_iter-num>1</Iteration_iter-num><Iteration_query-ID>Query_1</Iteration_query-ID>
<Iteration_query-def>gnl|alu|Z15030_HSAL001056 (Alu-J)</Iteration_query-def>
<Iteration_query-len>292</Iteration_query-len>
<Iteration_hits>
<Hit>
<Hit_num>1</Hit_num>
<Hit_id>gnl|alu|Z15030_HSAL001056</Hit_id>
<Hit_def>(Alu-J)</Hit_def>
<Hit_accession>Z15030_HSAL001056</Hit_accession>
<Hit_len>292</Hit_len>
<Hit_hsps>
<Hsp>
<Hsp_num>1</Hsp_num>
<Hsp_bit-score>540.342</Hsp_bit-score>
<Hsp_score>292</Hsp_score>
<Hsp_evalue>4.55414e-156</Hsp_evalue>
<Hsp_query-from>1</Hsp_query-from>
<Hsp_query-to>292</Hsp_query-to>
<Hsp_hit-from>1</Hsp_hit-from>
<Hsp_hit-to>292</Hsp_hit-to>
<Hsp_query-frame>1</Hsp_query-frame>
<Hsp_hit-frame>1</Hsp_hit-frame>
<Hsp_identity>292</Hsp_identity>
<Hsp_positive>292</Hsp_positive>
<Hsp_gaps>0</Hsp_gaps>
<Hsp_align-len>292</Hsp_align-len>
<Hsp_qseq>
AGGCTGGCACTGTGGCTCATGCTGAAATCCCAGCACGGCGGAGGACGGCGGAAGATTGCTTGAGCCTAGGAGTTTG
CGACCAGCCTGGGTGACATAGGGAGATGCCTGTCTCTACGCAAAAGAAAAAAAAAATAGCTCTGCTGGTGGTGCATG
CCTATAGTCTCAGCTATCAGGAGGCTGGGACAGGAGGATCACTTGGGCCCGGGAGTTGAGGCTGTGGTGAGCC
ACGATCACACCACTGCACTCCAGCCTGGGTGACAGAGCAAGACCCTGTCTCAAAACAAACAAATAA
</Hsp_qseq>
<Hsp_hseq>
AGGCTGGCACTGTGGCTCATGCTGAAATCCCAGCACGGCGGAGGACGGCGGAAGATTGCTTGAGCCTAGGA
GTTTGCGACCAGCCTGGGTGACATAGGGAGATGCCTGTCTCTACGCAAAAGAAAAAAAAAATAGCTCTGCT
GGTGGTGCATGCCTATAGTCTCAGCTATCAGGAGGCTGGGACAGGAGGATCACTTGGGCCCGGGAGTTGAGG
CTGTGGTGAGCCACGATCACACCACTGCACTCCAGCCTGGGTGACAGAGCAAGACCCTGTCTCAAAACAAAC
AAATAA
</Hsp_hseq>
<Hsp_midline>
|||||||||||||||||||||||||||||||||||||||||||||||||||||
|||||||||||||||||||||||||||||||||||||||||||||||||||||
|||||||||||||||||||||||||||||||||||||||||||||||||||||
|||||||||||||||||||||||||||||||||||||||||||||||||||||
|||||||||||||||||||||||||||||||||||||||||||||||||||||
|||||||||||||||||||||||||||
</Hsp_midline>
</Hsp>
</Hit_hsps>
</Hit>
.........................
.........................
.........................
</Iteration_hits>
<Iteration_stat>
<Statistics>
<Statistics_db-num>327</Statistics_db-num>
<Statistics_db-len>80506</Statistics_db-len>
<Statistics_hsp-lenv16</Statistics_hsp-len>
<Statistics_eff-space>21528364</Statistics_eff-space>
<Statistics_kappa>0.46</Statistics_kappa>
<Statistics_lambda>1.28</Statistics_lambda>
<Statistics_entropy>0.85</Statistics_entropy>
</Statistics>
</Iteration_stat>
</Iteration>
</BlastOutput_iterations>
</BlastOutput>
以上命令可以在python中使用以下程式碼執行:
>>> from Bio.Blast.Applications import NcbiblastnCommandline >>> blastn_cline = NcbiblastnCommandline(query = "search.fasta", db = "alun", outfmt = 5, out = "results.xml") >>> stdout, stderr = blastn_cline()
這裡,第一個是blast輸出的控制代碼,第二個是blast命令生成的可能的錯誤輸出。
由於我們已將輸出檔案作為命令列引數提供(out = “results.xml”)並將輸出格式設定為XML(outfmt = 5),因此輸出檔案將儲存在當前工作目錄中。
解析BLAST結果
通常,BLAST輸出使用NCBIXML模組解析為XML格式。為此,我們需要匯入以下模組:
>>> from Bio.Blast import NCBIXML
現在,使用python open方法直接開啟檔案並使用NCBIXML parse方法,如下所示:
>>> E_VALUE_THRESH = 1e-20
>>> for record in NCBIXML.parse(open("results.xml")):
>>> if record.alignments:
>>> print("\n")
>>> print("query: %s" % record.query[:100])
>>> for align in record.alignments:
>>> for hsp in align.hsps:
>>> if hsp.expect < E_VALUE_THRESH:
>>> print("match: %s " % align.title[:100])
這將產生如下輸出:
query: gnl|alu|Z15030_HSAL001056 (Alu-J) match: gnl|alu|Z15030_HSAL001056 (Alu-J) match: gnl|alu|L12964_HSAL003860 (Alu-J) match: gnl|alu|L13042_HSAL003863 (Alu-FLA?) match: gnl|alu|M86249_HSAL001462 (Alu-FLA?) match: gnl|alu|M29484_HSAL002265 (Alu-J) query: gnl|alu|D00596_HSAL003180 (Alu-Sx) match: gnl|alu|D00596_HSAL003180 (Alu-Sx) match: gnl|alu|J03071_HSAL001860 (Alu-J) match: gnl|alu|X72409_HSAL005025 (Alu-Sx) query: gnl|alu|X55502_HSAL000745 (Alu-J) match: gnl|alu|X55502_HSAL000745 (Alu-J)
Biopython - Entrez 資料庫
Entrez是NCBI提供的線上搜尋系統。它提供對幾乎所有已知的分子生物學資料庫的訪問,並集成了一個全域性查詢,支援布林運算子和欄位搜尋。它從所有資料庫返回結果,其中包含諸如每個資料庫的命中次數、帶有指向源資料庫連結的記錄等資訊。
一些可以透過Entrez訪問的流行資料庫列在下面:
- PubMed
- PubMed Central
- 核苷酸(GenBank序列資料庫)
- 蛋白質(序列資料庫)
- 基因組(全基因組資料庫)
- 結構(三維大分子結構)
- 分類(GenBank中的生物體)
- SNP(單核苷酸多型性)
- UniGene(轉錄序列的基因導向簇)
- CDD(保守蛋白結構域資料庫)
- 3D結構域(來自Entrez結構的結構域)
除了上述資料庫之外,Entrez 還提供了更多資料庫來執行欄位搜尋。
Biopython 提供了一個 Entrez 特定的模組 Bio.Entrez 來訪問 Entrez 資料庫。讓我們在本節中學習如何使用 Biopython 訪問 Entrez -
資料庫連線步驟
要新增 Entrez 的功能,請匯入以下模組 -
>>> from Bio import Entrez
接下來設定您的電子郵件以識別誰連線到下面給出的程式碼 -
>>> Entrez.email = '<youremail>'
然後,設定 Entrez 工具引數,預設情況下為 Biopython。
>>> Entrez.tool = 'Demoscript'
現在,呼叫 einfo 函式以查詢每個資料庫的索引詞計數、上次更新和可用連結,如下所示 -
>>> info = Entrez.einfo()
einfo 方法返回一個物件,該物件透過其 read 方法提供對資訊的訪問,如下所示 -
>>> data = info.read()
>>> print(data)
<?xml version = "1.0" encoding = "UTF-8" ?>
<!DOCTYPE eInfoResult PUBLIC "-//NLM//DTD einfo 20130322//EN"
"https://eutils.ncbi.nlm.nih.gov/eutils/dtd/20130322/einfo.dtd">
<eInfoResult>
<DbList>
<DbName>pubmed</DbName>
<DbName>protein</DbName>
<DbName>nuccore</DbName>
<DbName>ipg</DbName>
<DbName>nucleotide</DbName>
<DbName>nucgss</DbName>
<DbName>nucest</DbName>
<DbName>structure</DbName>
<DbName>sparcle</DbName>
<DbName>genome</DbName>
<DbName>annotinfo</DbName>
<DbName>assembly</DbName>
<DbName>bioproject</DbName>
<DbName>biosample</DbName>
<DbName>blastdbinfo</DbName>
<DbName>books</DbName>
<DbName>cdd</DbName>
<DbName>clinvar</DbName>
<DbName>clone</DbName>
<DbName>gap</DbName>
<DbName>gapplus</DbName>
<DbName>grasp</DbName>
<DbName>dbvar</DbName>
<DbName>gene</DbName>
<DbName>gds</DbName>
<DbName>geoprofiles</DbName>
<DbName>homologene</DbName>
<DbName>medgen</DbName>
<DbName>mesh</DbName>
<DbName>ncbisearch</DbName>
<DbName>nlmcatalog</DbName>
<DbName>omim</DbName>
<DbName>orgtrack</DbName>
<DbName>pmc</DbName>
<DbName>popset</DbName>
<DbName>probe</DbName>
<DbName>proteinclusters</DbName>
<DbName>pcassay</DbName>
<DbName>biosystems</DbName>
<DbName>pccompound</DbName>
<DbName>pcsubstance</DbName>
<DbName>pubmedhealth</DbName>
<DbName>seqannot</DbName>
<DbName>snp</DbName>
<DbName>sra</DbName>
<DbName>taxonomy</DbName>
<DbName>biocollections</DbName>
<DbName>unigene</DbName>
<DbName>gencoll</DbName>
<DbName>gtr</DbName>
</DbList>
</eInfoResult>
資料採用 XML 格式,要將資料作為 python 物件獲取,請在呼叫Entrez.einfo()方法後立即使用Entrez.read方法 -
>>> info = Entrez.einfo() >>> record = Entrez.read(info)
這裡,record 是一個字典,它有一個鍵 DbList,如下所示 -
>>> record.keys() [u'DbList']
訪問 DbList 鍵將返回資料庫名稱列表,如下所示 -
>>> record[u'DbList'] ['pubmed', 'protein', 'nuccore', 'ipg', 'nucleotide', 'nucgss', 'nucest', 'structure', 'sparcle', 'genome', 'annotinfo', 'assembly', 'bioproject', 'biosample', 'blastdbinfo', 'books', 'cdd', 'clinvar', 'clone', 'gap', 'gapplus', 'grasp', 'dbvar', 'gene', 'gds', 'geoprofiles', 'homologene', 'medgen', 'mesh', 'ncbisearch', 'nlmcatalog', 'omim', 'orgtrack', 'pmc', 'popset', 'probe', 'proteinclusters', 'pcassay', 'biosystems', 'pccompound', 'pcsubstance', 'pubmedhealth', 'seqannot', 'snp', 'sra', 'taxonomy', 'biocollections', 'unigene', 'gencoll', 'gtr'] >>>
基本上,Entrez 模組解析 Entrez 搜尋系統返回的 XML 並將其作為 python 字典和列表提供。
搜尋資料庫
要搜尋任何一個 Entrez 資料庫,我們可以使用 Bio.Entrez.esearch() 模組。它定義如下 -
>>> info = Entrez.einfo()
>>> info = Entrez.esearch(db = "pubmed",term = "genome")
>>> record = Entrez.read(info)
>>>print(record)
DictElement({u'Count': '1146113', u'RetMax': '20', u'IdList':
['30347444', '30347404', '30347317', '30347292',
'30347286', '30347249', '30347194', '30347187',
'30347172', '30347088', '30347075', '30346992',
'30346990', '30346982', '30346980', '30346969',
'30346962', '30346954', '30346941', '30346939'],
u'TranslationStack': [DictElement({u'Count':
'927819', u'Field': 'MeSH Terms', u'Term': '"genome"[MeSH Terms]',
u'Explode': 'Y'}, attributes = {})
, DictElement({u'Count': '422712', u'Field':
'All Fields', u'Term': '"genome"[All Fields]', u'Explode': 'N'}, attributes = {}),
'OR', 'GROUP'], u'TranslationSet': [DictElement({u'To': '"genome"[MeSH Terms]
OR "genome"[All Fields]', u'From': 'genome'}, attributes = {})], u'RetStart': '0',
u'QueryTranslation': '"genome"[MeSH Terms] OR "genome"[All Fields]'},
attributes = {})
>>>
如果您分配了不正確的資料庫,則它將返回
>>> info = Entrez.esearch(db = "blastdbinfo",term = "books")
>>> record = Entrez.read(info)
>>> print(record)
DictElement({u'Count': '0', u'RetMax': '0', u'IdList': [],
u'WarningList': DictElement({u'OutputMessage': ['No items found.'],
u'PhraseIgnored': [], u'QuotedPhraseNotFound': []}, attributes = {}),
u'ErrorList': DictElement({u'FieldNotFound': [], u'PhraseNotFound':
['books']}, attributes = {}), u'TranslationSet': [], u'RetStart': '0',
u'QueryTranslation': '(books[All Fields])'}, attributes = {})
如果要跨資料庫搜尋,則可以使用Entrez.egquery。這類似於Entrez.esearch,只是它只需指定關鍵字並跳過資料庫引數即可。
>>>info = Entrez.egquery(term = "entrez") >>> record = Entrez.read(info) >>> for row in record["eGQueryResult"]: ... print(row["DbName"], row["Count"]) ... pubmed 458 pmc 12779 mesh 1 ... ... ... biosample 7 biocollections 0
獲取記錄
Enterz 提供了一個特殊的方法 efetch 來搜尋和下載 Entrez 中記錄的完整詳細資訊。考慮以下簡單示例 -
>>> handle = Entrez.efetch( db = "nucleotide", id = "EU490707", rettype = "fasta")
現在,我們可以簡單地使用 SeqIO 物件讀取記錄
>>> record = SeqIO.read( handle, "fasta" )
>>> record
SeqRecord(seq = Seq('ATTTTTTACGAACCTGTGGAAATTTTTGGTTATGACAATAAATCTAGTTTAGTA...GAA',
SingleLetterAlphabet()), id = 'EU490707.1', name = 'EU490707.1',
description = 'EU490707.1
Selenipedium aequinoctiale maturase K (matK) gene, partial cds; chloroplast',
dbxrefs = [])
Biopython - PDB 模組
Biopython 提供 Bio.PDB 模組來處理多肽結構。PDB(蛋白質資料庫)是可線上獲取的最大蛋白質結構資源。它承載了許多不同的蛋白質結構,包括蛋白質-蛋白質、蛋白質-DNA、蛋白質-RNA 複合物。
為了載入 PDB,請鍵入以下命令 -
from Bio.PDB import *
蛋白質結構檔案格式
PDB 以三種不同的格式分發蛋白質結構 -
- 基於 XML 的檔案格式,Biopython 不支援該格式
- pdb 檔案格式,這是一種特殊格式的文字檔案
- PDBx/mmCIF 檔案格式
蛋白質資料庫分發的 PDB 檔案可能包含格式錯誤,這些錯誤會使它們變得模稜兩可或難以解析。Bio.PDB 模組嘗試自動處理這些錯誤。
Bio.PDB 模組實現了兩個不同的解析器,一個是 mmCIF 格式,另一個是 pdb 格式。
讓我們詳細瞭解如何解析每種格式 -
mmCIF 解析器
讓我們使用以下命令從 pdb 伺服器下載 mmCIF 格式的示例資料庫 -
>>> pdbl = PDBList()
>>> pdbl.retrieve_pdb_file('2FAT', pdir = '.', file_format = 'mmCif')
這將從伺服器下載指定的檔案 (2fat.cif) 並將其儲存在當前工作目錄中。
這裡,PDBList 提供了列出和下載來自線上 PDB FTP 伺服器的檔案的選項。retrieve_pdb_file 方法需要要下載的檔案的名稱,不帶副檔名。retrieve_pdb_file 還可以選擇指定下載目錄 pdir 和檔案格式 file_format。檔案格式的可能值為:-
- “mmCif”(預設,PDBx/mmCif 檔案)
- “pdb”(格式 PDB)
- “xml”(PMDML/XML 格式)
- “mmtf”(高度壓縮)
- “bundle”(用於大型結構的 PDB 格式存檔)
要載入 cif 檔案,請使用 Bio.MMCIF.MMCIFParser,如下所示 -
>>> parser = MMCIFParser(QUIET = True)
>>> data = parser.get_structure("2FAT", "2FAT.cif")
這裡,QUIET 在解析檔案期間抑制警告。get_structure 將解析檔案並返回結構,其 ID 為 2FAT(第一個引數)。
執行上述命令後,它將解析檔案並列印可能的警告(如果可用)。
現在,使用以下命令檢查結構 -
>>> data <Structure id = 2FAT> To get the type, use type method as specified below, >>> print(type(data)) <class 'Bio.PDB.Structure.Structure'>
我們已成功解析檔案並獲得了蛋白質的結構。我們將在後面的章節中學習蛋白質結構的詳細資訊以及如何獲取它。
PDB 解析器
讓我們使用以下命令從 pdb 伺服器下載 PDB 格式的示例資料庫 -
>>> pdbl = PDBList()
>>> pdbl.retrieve_pdb_file('2FAT', pdir = '.', file_format = 'pdb')
這將從伺服器下載指定的檔案 (pdb2fat.ent) 並將其儲存在當前工作目錄中。
要載入 pdb 檔案,請使用 Bio.PDB.PDBParser,如下所示 -
>>> parser = PDBParser(PERMISSIVE = True, QUIET = True)
>>> data = parser.get_structure("2fat","pdb2fat.ent")
這裡,get_structure 類似於 MMCIFParser。PERMISSIVE 選項嘗試儘可能靈活地解析蛋白質資料。
現在,使用下面給出的程式碼片段檢查結構及其型別 -
>>> data <Structure id = 2fat> >>> print(type(data)) <class 'Bio.PDB.Structure.Structure'>
好吧,標題結構儲存字典資訊。為此,請鍵入以下命令 -
>>> print(data.header.keys()) dict_keys([ 'name', 'head', 'deposition_date', 'release_date', 'structure_method', 'resolution', 'structure_reference', 'journal_reference', 'author', 'compound', 'source', 'keywords', 'journal']) >>>
要獲取名稱,請使用以下程式碼 -
>>> print(data.header["name"]) an anti-urokinase plasminogen activator receptor (upar) antibody: crystal structure and binding epitope >>>
您還可以使用以下程式碼檢查日期和解析度 -
>>> print(data.header["release_date"]) 2006-11-14 >>> print(data.header["resolution"]) 1.77
PDB 結構
PDB 結構由單個模型組成,包含兩條鏈。
- 鏈 L,包含多個殘基
- 鏈 H,包含多個殘基
每個殘基由多個原子組成,每個原子都有一個 3D 位置,由 (x、y、z) 座標表示。
讓我們在下一節中詳細瞭解如何獲取原子的結構 -
模型
Structure.get_models() 方法返回模型的迭代器。它定義如下 -
>>> model = data.get_models() >>> model <generator object get_models at 0x103fa1c80> >>> models = list(model) >>> models [<Model id = 0>] >>> type(models[0]) <class 'Bio.PDB.Model.Model'>
這裡,Model 精確描述了一種 3D 構象。它包含一個或多個鏈。
鏈
Model.get_chain() 方法返回鏈的迭代器。它定義如下 -
>>> chains = list(models[0].get_chains()) >>> chains [<Chain id = L>, <Chain id = H>] >>> type(chains[0]) <class 'Bio.PDB.Chain.Chain'>
這裡,Chain 描述了一個正確的多肽結構,即一系列連續結合的殘基。
殘基
Chain.get_residues() 方法返回殘基的迭代器。它定義如下 -
>>> residue = list(chains[0].get_residues()) >>> len(residue) 293 >>> residue1 = list(chains[1].get_residues()) >>> len(residue1) 311
好吧,Residue 儲存屬於氨基酸的原子。
原子
Residue.get_atom() 返回原子的迭代器,定義如下 -
>>> atoms = list(residue[0].get_atoms()) >>> atoms [<Atom N>, <Atom CA>, <Atom C>, <Atom Ov, <Atom CB>, <Atom CG>, <Atom OD1>, <Atom OD2>]
原子儲存原子的 3D 座標,稱為向量。它定義如下
>>> atoms[0].get_vector() <Vector 18.49, 73.26, 44.16>
它表示 x、y 和 z 座標值。
Biopython - 基序物件
序列基序是核苷酸或氨基酸序列模式。序列基序由可能不相鄰的氨基酸的三維排列形成。Biopython 提供了一個單獨的模組 Bio.motifs 來訪問序列基序的功能,如下所示 -
from Bio import motifs
建立簡單的 DNA 基序
讓我們使用以下命令建立一個簡單的 DNA 基序序列 -
>>> from Bio import motifs
>>> from Bio.Seq import Seq
>>> DNA_motif = [ Seq("AGCT"),
... Seq("TCGA"),
... Seq("AACT"),
... ]
>>> seq = motifs.create(DNA_motif)
>>> print(seq) AGCT TCGA AACT
要計算序列值,請使用以下命令 -
>>> print(seq.counts)
0 1 2 3
A: 2.00 1.00 0.00 1.00
C: 0.00 1.00 2.00 0.00
G: 0.00 1.00 1.00 0.00
T: 1.00 0.00 0.00 2.00
使用以下程式碼計算序列中的“A” -
>>> seq.counts["A", :] (2, 1, 0, 1)
如果要訪問計數列,請使用以下命令 -
>>> seq.counts[:, 3]
{'A': 1, 'C': 0, 'T': 2, 'G': 0}
建立序列標識
我們現在將討論如何建立序列標識。
考慮以下序列 -
AGCTTACG ATCGTACC TTCCGAAT GGTACGTA AAGCTTGG
您可以使用以下連結建立自己的標識:http://weblogo.berkeley.edu/
新增上述序列並建立一個新的標識,並將名為 seq.png 的影像儲存到您的 biopython 資料夾中。
seq.png

建立影像後,現在執行以下命令 -
>>> seq.weblogo("seq.png")
此 DNA 序列基序表示為 LexA 結合基序的序列標識。
JASPAR 資料庫
JASPAR 是最流行的資料庫之一。它為任何基序格式提供讀取、寫入和掃描序列的功能。它儲存每個基序的元資訊。Bio.motifs 模組包含一個專門的類 jaspar.Motif 來表示元資訊屬性。
它具有以下值得注意的屬性型別 -
- matrix_id - 唯一的 JASPAR 基序 ID
- name - 基序的名稱
- tf_family - 基序的家族,例如“螺旋-環-螺旋”
- data_type - 基序中使用的資料型別。
讓我們在 biopython 資料夾中建立一個名為 sample.sites 的 JASPAR 站點格式。它定義如下 -
sample.sites >MA0001 ARNT 1 AACGTGatgtccta >MA0001 ARNT 2 CAGGTGggatgtac >MA0001 ARNT 3 TACGTAgctcatgc >MA0001 ARNT 4 AACGTGacagcgct >MA0001 ARNT 5 CACGTGcacgtcgt >MA0001 ARNT 6 cggcctCGCGTGc
在上面的檔案中,我們建立了基序例項。現在,讓我們從上述例項建立一個基序物件 -
>>> from Bio import motifs
>>> with open("sample.sites") as handle:
... data = motifs.read(handle,"sites")
...
>>> print(data)
TF name None
Matrix ID None
Matrix:
0 1 2 3 4 5
A: 2.00 5.00 0.00 0.00 0.00 1.00
C: 3.00 0.00 5.00 0.00 0.00 0.00
G: 0.00 1.00 1.00 6.00 0.00 5.00
T: 1.00 0.00 0.00 0.00 6.00 0.00
這裡,data 從 sample.sites 檔案讀取所有基序例項。
要列印 data 中的所有例項,請使用以下命令 -
>>> for instance in data.instances: ... print(instance) ... AACGTG CAGGTG TACGTA AACGTG CACGTG CGCGTG
使用以下命令計算所有值 -
>>> print(data.counts)
0 1 2 3 4 5
A: 2.00 5.00 0.00 0.00 0.00 1.00
C: 3.00 0.00 5.00 0.00 0.00 0.00
G: 0.00 1.00 1.00 6.00 0.00 5.00
T: 1.00 0.00 0.00 0.00 6.00 0.00
>>>
Biopython - BioSQL 模組
BioSQL 是一種通用資料庫模式,主要設計用於儲存序列及其所有 RDBMS 引擎的相關資料。它的設計方式使其能夠儲存來自所有流行的生物資訊學資料庫(如 GenBank、Swissport 等)的資料。它也可以用於儲存內部資料。
BioSQL 目前為以下資料庫提供特定的模式 -
- MySQL (biosqldb-mysql.sql)
- PostgreSQL (biosqldb-pg.sql)
- Oracle (biosqldb-ora/*.sql)
- SQLite (biosqldb-sqlite.sql)
它還為基於 Java 的 HSQLDB 和 Derby 資料庫提供最少的支援。
BioPython 提供非常簡單、易用和高階的 ORM 功能來處理基於 BioSQL 的資料庫。BioPython 提供了一個模組 BioSQL 來執行以下功能 -
- 建立/刪除 BioSQL 資料庫
- 連線到 BioSQL 資料庫
- 解析序列資料庫(如 GenBank、Swisport、BLAST 結果、Entrez 結果等),並將其直接載入到 BioSQL 資料庫中
- 從 BioSQL 資料庫中獲取序列資料
- 從 NCBI BLAST 中獲取分類資料並將其儲存到 BioSQL 資料庫中
- 對 BioSQL 資料庫執行任何 SQL 查詢
BioSQL 資料庫模式概述
在深入瞭解 BioSQL 之前,讓我們瞭解 BioSQL 模式的基礎知識。BioSQL 模式提供 25 個以上的表來儲存序列資料、序列特徵、序列類別/本體和分類資訊。一些重要的表如下 -
- biodatabase
- bioentry
- biosequence
- seqfeature
- taxon
- taxon_name
- antology
- term
- dxref
建立 BioSQL 資料庫
在本節中,讓我們使用 BioSQL 團隊提供的模式建立一個示例 BioSQL 資料庫 biosql。我們將使用 SQLite 資料庫,因為它非常易於上手並且沒有複雜的設定。
這裡,我們將使用以下步驟建立一個基於 SQLite 的 BioSQL 資料庫。
步驟 1 - 下載 SQLite 資料庫引擎並安裝它。
步驟 2 - 從 GitHub URL 下載 BioSQL 專案。https://github.com/biosql/biosql
步驟 3 - 開啟控制檯並使用 mkdir 建立一個目錄並進入該目錄。
cd /path/to/your/biopython/sample mkdir sqlite-biosql cd sqlite-biosql
步驟 4 - 執行以下命令以建立一個新的 SQLite 資料庫。
> sqlite3.exe mybiosql.db SQLite version 3.25.2 2018-09-25 19:08:10 Enter ".help" for usage hints. sqlite>
步驟 5 - 從 BioSQL 專案複製 biosqldb-sqlite.sql 檔案(/sql/biosqldb-sqlite.sql`)並將其儲存在當前目錄中。
步驟 6 - 執行以下命令以建立所有表。
sqlite> .read biosqldb-sqlite.sql
現在,所有表都已建立到我們的新資料庫中。
步驟 7 - 執行以下命令以檢視資料庫中的所有新表。
sqlite> .headers on sqlite> .mode column sqlite> .separator ROW "\n" sqlite> SELECT name FROM sqlite_master WHERE type = 'table'; biodatabase taxon taxon_name ontology term term_synonym term_dbxref term_relationship term_relationship_term term_path bioentry bioentry_relationship bioentry_path biosequence dbxref dbxref_qualifier_value bioentry_dbxref reference bioentry_reference comment bioentry_qualifier_value seqfeature seqfeature_relationship seqfeature_path seqfeature_qualifier_value seqfeature_dbxref location location_qualifier_value sqlite>
前三個命令是配置命令,用於配置 SQLite 以格式化的方式顯示結果。
步驟 8 - 將 BioPython 團隊提供的示例 GenBank 檔案 ls_orchid.gbk 複製到當前目錄https://raw.githubusercontent.com/biopython/biopython/master/Doc/examples/ls_orchid.gbk並將其另存為 orchid.gbk。
步驟 9 - 使用以下程式碼建立一個 python 指令碼 load_orchid.py 並執行它。
from Bio import SeqIO
from BioSQL import BioSeqDatabase
import os
server = BioSeqDatabase.open_database(driver = 'sqlite3', db = "orchid.db")
db = server.new_database("orchid")
count = db.load(SeqIO.parse("orchid.gbk", "gb"), True) server.commit()
server.close()
以上程式碼解析檔案中的記錄並將其轉換為 python 物件,然後將其插入到 BioSQL 資料庫中。我們將在後面的章節中分析程式碼。
最後,我們建立了一個新的 BioSQL 資料庫並將一些示例資料載入到其中。我們將在下一章討論重要的表。
簡單的 ER 圖
biodatabase 表位於層次結構的頂部,其主要目的是將一組序列資料組織到單個組/虛擬資料庫中。biodatabase 中的每個條目都指的是一個單獨的資料庫,並且不會與其他資料庫混合。BioSQL 資料庫中的所有相關表都引用 biodatabase 條目。
bioentry 表儲存序列的所有詳細資訊,但序列資料除外。特定bioentry的序列資料將儲存在biosequence表中。
taxon 和 taxon_name 是分類詳細資訊,每個條目都引用此表以指定其分類資訊。
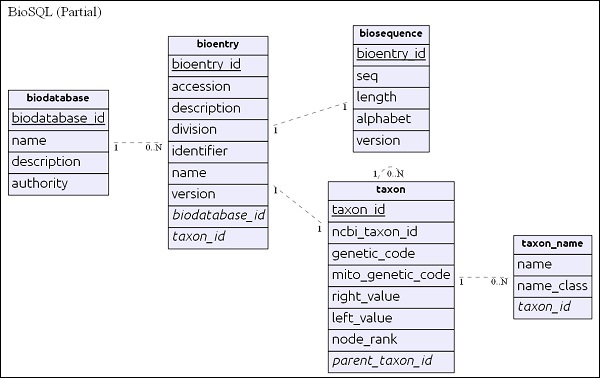
在瞭解了模式後,讓我們在下一節中檢視一些查詢。
BioSQL 查詢
讓我們深入研究一些SQL查詢,以便更好地理解資料是如何組織的以及表是如何相互關聯的。在繼續之前,讓我們使用以下命令開啟資料庫並設定一些格式化命令:
> sqlite3 orchid.db SQLite version 3.25.2 2018-09-25 19:08:10 Enter ".help" for usage hints. sqlite> .header on sqlite> .mode columns
.header 和 .mode 是格式化選項,可以更好地視覺化資料。您也可以使用任何SQLite編輯器來執行查詢。
列出系統中可用的虛擬序列資料庫,如下所示:
select * from biodatabase; *** Result *** sqlite> .width 15 15 15 15 sqlite> select * from biodatabase; biodatabase_id name authority description --------------- --------------- --------------- --------------- 1 orchid sqlite>
這裡,我們只有一個數據庫,orchid。
列出資料庫orchid中可用的條目(前3個),使用以下程式碼
select
be.*,
bd.name
from
bioentry be
inner join
biodatabase bd
on bd.biodatabase_id = be.biodatabase_id
where
bd.name = 'orchid' Limit 1,
3;
*** Result ***
sqlite> .width 15 15 10 10 10 10 10 50 10 10
sqlite> select be.*, bd.name from bioentry be inner join biodatabase bd on
bd.biodatabase_id = be.biodatabase_id where bd.name = 'orchid' Limit 1,3;
bioentry_id biodatabase_id taxon_id name accession identifier division description version name
--------------- --------------- ---------- ---------- ---------- ---------- ----------
---------- ---------- ----------- ---------- --------- ---------- ----------
2 1 19 Z78532 Z78532 2765657 PLN
C.californicum 5.8S rRNA gene and ITS1 and ITS2 DN 1
orchid
3 1 20 Z78531 Z78531 2765656 PLN
C.fasciculatum 5.8S rRNA gene and ITS1 and ITS2 DN 1
orchid
4 1 21 Z78530 Z78530 2765655 PLN
C.margaritaceum 5.8S rRNA gene and ITS1 and ITS2 D 1
orchid
sqlite>
列出與一個條目(登入號 - Z78530,名稱 - C. fasciculatum 5.8S rRNA基因和ITS1和ITS2 DNA)關聯的序列詳細資訊,使用以下程式碼:
select
substr(cast(bs.seq as varchar), 0, 10) || '...' as seq,
bs.length,
be.accession,
be.description,
bd.name
from
biosequence bs
inner join
bioentry be
on be.bioentry_id = bs.bioentry_id
inner join
biodatabase bd
on bd.biodatabase_id = be.biodatabase_id
where
bd.name = 'orchid'
and be.accession = 'Z78532';
*** Result ***
sqlite> .width 15 5 10 50 10
sqlite> select substr(cast(bs.seq as varchar), 0, 10) || '...' as seq,
bs.length, be.accession, be.description, bd.name from biosequence bs inner
join bioentry be on be.bioentry_id = bs.bioentry_id inner join biodatabase bd
on bd.biodatabase_id = be.biodatabase_id where bd.name = 'orchid' and
be.accession = 'Z78532';
seq length accession description name
------------ ---------- ---------- ------------ ------------ ---------- ---------- -----------------
CGTAACAAG... 753 Z78532 C.californicum 5.8S rRNA gene and ITS1 and ITS2 DNA orchid
sqlite>
使用以下程式碼獲取與一個條目(登入號 - Z78530,名稱 - C. fasciculatum 5.8S rRNA基因和ITS1和ITS2 DNA)關聯的完整序列:
select
bs.seq
from
biosequence bs
inner join
bioentry be
on be.bioentry_id = bs.bioentry_id
inner join
biodatabase bd
on bd.biodatabase_id = be.biodatabase_id
where
bd.name = 'orchid'
and be.accession = 'Z78532';
*** Result ***
sqlite> .width 1000
sqlite> select bs.seq from biosequence bs inner join bioentry be on
be.bioentry_id = bs.bioentry_id inner join biodatabase bd on bd.biodatabase_id =
be.biodatabase_id where bd.name = 'orchid' and be.accession = 'Z78532';
seq
----------------------------------------------------------------------------------------
----------------------------
CGTAACAAGGTTTCCGTAGGTGAACCTGCGGAAGGATCATTGTTGAGACAACAGAATATATGATCGAGTGAATCT
GGAGGACCTGTGGTAACTCAGCTCGTCGTGGCACTGCTTTTGTCGTGACCCTGCTTTGTTGTTGGGCCTCC
TCAAGAGCTTTCATGGCAGGTTTGAACTTTAGTACGGTGCAGTTTGCGCCAAGTCATATAAAGCATCACTGATGAATGACATTATTGT
CAGAAAAAATCAGAGGGGCAGTATGCTACTGAGCATGCCAGTGAATTTTTATGACTCTCGCAACGGATATCTTGGCTC
TAACATCGATGAAGAACGCAG
sqlite>
列出與生物資料庫orchid相關的分類群
select distinct
tn.name
from
biodatabase d
inner join
bioentry e
on e.biodatabase_id = d.biodatabase_id
inner join
taxon t
on t.taxon_id = e.taxon_id
inner join
taxon_name tn
on tn.taxon_id = t.taxon_id
where
d.name = 'orchid' limit 10;
*** Result ***
sqlite> select distinct tn.name from biodatabase d inner join bioentry e on
e.biodatabase_id = d.biodatabase_id inner join taxon t on t.taxon_id =
e.taxon_id inner join taxon_name tn on tn.taxon_id = t.taxon_id where d.name =
'orchid' limit 10;
name
------------------------------
Cypripedium irapeanum
Cypripedium californicum
Cypripedium fasciculatum
Cypripedium margaritaceum
Cypripedium lichiangense
Cypripedium yatabeanum
Cypripedium guttatum
Cypripedium acaule
pink lady's slipper
Cypripedium formosanum
sqlite>
將資料載入到BioSQL資料庫中
讓我們在本節中學習如何將序列資料載入到BioSQL資料庫中。我們已經在上一節中提供了將資料載入到資料庫中的程式碼,程式碼如下:
from Bio import SeqIO
from BioSQL import BioSeqDatabase
import os
server = BioSeqDatabase.open_database(driver = 'sqlite3', db = "orchid.db")
DBSCHEMA = "biosqldb-sqlite.sql"
SQL_FILE = os.path.join(os.getcwd(), DBSCHEMA)
server.load_database_sql(SQL_FILE)
server.commit()
db = server.new_database("orchid")
count = db.load(SeqIO.parse("orchid.gbk", "gb"), True) server.commit()
server.close()
我們將深入瞭解程式碼的每一行及其用途:
第1行 - 載入SeqIO模組。
第2行 - 載入BioSeqDatabase模組。此模組提供與BioSQL資料庫互動的所有功能。
第3行 - 載入os模組。
第5行 - open_database開啟指定資料庫(db),使用配置的驅動程式(driver),並返回到BioSQL資料庫(server)的控制代碼。Biopython支援sqlite、mysql、postgresql和oracle資料庫。
第6-10行 - load_database_sql方法載入外部檔案中的sql並執行它。commit方法提交事務。我們可以跳過此步驟,因為我們已經建立了具有模式的資料庫。
第12行 - new_database方法建立新的虛擬資料庫orchid,並返回一個控制代碼db以針對orchid資料庫執行命令。
第13行 - load方法將序列條目(可迭代的SeqRecord)載入到orchid資料庫中。SqlIO.parse解析GenBank資料庫,並將其中的所有序列作為可迭代的SeqRecord返回。load方法的第二個引數(True)指示它從NCBI blast網站獲取序列資料的分類學詳細資訊,如果系統中尚不存在。
第14行 - commit提交事務。
第15行 - close關閉資料庫連線並銷燬伺服器控制代碼。
獲取序列資料
讓我們從orchid資料庫中獲取識別符號為2765658的序列,如下所示:
from BioSQL import BioSeqDatabase
server = BioSeqDatabase.open_database(driver = 'sqlite3', db = "orchid.db")
db = server["orchid"]
seq_record = db.lookup(gi = 2765658)
print(seq_record.id, seq_record.description[:50] + "...")
print("Sequence length %i," % len(seq_record.seq))
這裡,server["orchid"]返回控制代碼以從虛擬資料庫orchid中獲取資料。lookup方法提供了一個根據條件選擇序列的選項,我們選擇了識別符號為2765658的序列。lookup將序列資訊作為SeqRecord物件返回。由於我們已經知道如何使用SeqRecord,因此很容易從中獲取資料。
刪除資料庫
刪除資料庫就像使用正確的資料庫名稱呼叫remove_database方法然後提交它一樣簡單,如下所示:
from BioSQL import BioSeqDatabase
server = BioSeqDatabase.open_database(driver = 'sqlite3', db = "orchid.db")
server.remove_database("orchids")
server.commit()
Biopython - 群體遺傳學
群體遺傳學在進化理論中發揮著重要作用。它分析物種之間以及同一物種內兩個或多個個體之間的遺傳差異。
Biopython為群體遺傳學提供了Bio.PopGen模組,主要支援`GenePop,這是Michel Raymond和Francois Rousset開發的一個流行的遺傳學軟體包。
一個簡單的解析器
讓我們編寫一個簡單的應用程式來解析GenePop格式並理解其概念。
下載Biopython團隊在以下連結中提供的genePop檔案:
https://raw.githubusercontent.com/biopython/biopython/master/Tests/PopGen/c3line.gen使用以下程式碼片段載入GenePop模組:
from Bio.PopGen import GenePop
使用GenePop.read方法解析檔案,如下所示:
record = GenePop.read(open("c3line.gen"))
顯示基因座和種群資訊,如下所示:
>>> record.loci_list
['136255903', '136257048', '136257636']
>>> record.pop_list
['4', 'b3', '5']
>>> record.populations
[[('1', [(3, 3), (4, 4), (2, 2)]), ('2', [(3, 3), (3, 4), (2, 2)]),
('3', [(3, 3), (4, 4), (2, 2)]), ('4', [(3, 3), (4, 3), (None, None)])],
[('b1', [(None, None), (4, 4), (2, 2)]), ('b2', [(None, None), (4, 4), (2, 2)]),
('b3', [(None, None), (4, 4), (2, 2)])],
[('1', [(3, 3), (4, 4), (2, 2)]), ('2', [(3, 3), (1, 4), (2, 2)]),
('3', [(3, 2), (1, 1), (2, 2)]), ('4',
[(None, None), (4, 4), (2, 2)]), ('5', [(3, 3), (4, 4), (2, 2)])]]
>>>
這裡,檔案中存在三個基因座和三組種群:第一組種群有4條記錄,第二組種群有3條記錄,第三組種群有5條記錄。record.populations顯示所有種群集,每個基因座的等位基因資料。
操作GenePop檔案
Biopython提供刪除基因座和種群資料的選項。
按位置刪除種群集,
>>> record.remove_population(0)
>>> record.populations
[[('b1', [(None, None), (4, 4), (2, 2)]),
('b2', [(None, None), (4, 4), (2, 2)]),
('b3', [(None, None), (4, 4), (2, 2)])],
[('1', [(3, 3), (4, 4), (2, 2)]),
('2', [(3, 3), (1, 4), (2, 2)]),
('3', [(3, 2), (1, 1), (2, 2)]),
('4', [(None, None), (4, 4), (2, 2)]),
('5', [(3, 3), (4, 4), (2, 2)])]]
>>>
按位置刪除基因座,
>>> record.remove_locus_by_position(0)
>>> record.loci_list
['136257048', '136257636']
>>> record.populations
[[('b1', [(4, 4), (2, 2)]), ('b2', [(4, 4), (2, 2)]), ('b3', [(4, 4), (2, 2)])],
[('1', [(4, 4), (2, 2)]), ('2', [(1, 4), (2, 2)]),
('3', [(1, 1), (2, 2)]), ('4', [(4, 4), (2, 2)]), ('5', [(4, 4), (2, 2)])]]
>>>
按名稱刪除基因座,
>>> record.remove_locus_by_name('136257636') >>> record.loci_list
['136257048']
>>> record.populations
[[('b1', [(4, 4)]), ('b2', [(4, 4)]), ('b3', [(4, 4)])],
[('1', [(4, 4)]), ('2', [(1, 4)]),
('3', [(1, 1)]), ('4', [(4, 4)]), ('5', [(4, 4)])]]
>>>
與GenePop軟體互動
Biopython提供與GenePop軟體互動的介面,從而公開其中的許多功能。Bio.PopGen.GenePop模組用於此目的。一個易於使用的介面是EasyController。讓我們檢查如何解析GenePop檔案並使用EasyController進行一些分析。
首先,安裝GenePop軟體並將安裝資料夾放在系統路徑中。要獲取有關GenePop檔案的基本資訊,請建立一個EasyController物件,然後呼叫get_basic_info方法,如下所示:
>>> from Bio.PopGen.GenePop.EasyController import EasyController
>>> ec = EasyController('c3line.gen')
>>> print(ec.get_basic_info())
(['4', 'b3', '5'], ['136255903', '136257048', '136257636'])
>>>
這裡,第一項是種群列表,第二項是基因座列表。
要獲取特定基因座的所有等位基因列表,請透過傳遞基因座名稱來呼叫get_alleles_all_pops方法,如下所示:
>>> allele_list = ec.get_alleles_all_pops("136255903")
>>> print(allele_list)
[2, 3]
要獲取特定種群和基因座的等位基因列表,請透過傳遞基因座名稱和種群位置來呼叫get_alleles方法,如下所示:
>>> allele_list = ec.get_alleles(0, "136255903") >>> print(allele_list) [] >>> allele_list = ec.get_alleles(1, "136255903") >>> print(allele_list) [] >>> allele_list = ec.get_alleles(2, "136255903") >>> print(allele_list) [2, 3] >>>
類似地,EasyController公開了許多功能:等位基因頻率、基因型頻率、多基因座F統計量、Hardy-Weinberg平衡、連鎖不平衡等。
Biopython - 基因組分析
基因組是完整的DNA集,包括其所有基因。基因組分析是指研究單個基因及其在遺傳中的作用。
基因組圖
基因組圖將遺傳資訊表示為圖表。Biopython使用Bio.Graphics.GenomeDiagram模組來表示GenomeDiagram。GenomeDiagram模組需要安裝ReportLab。
建立圖表的步驟
建立圖表的流程通常遵循以下簡單模式:
為要顯示的每個單獨的特徵集建立一個FeatureSet,並將Bio.SeqFeature物件新增到其中。
為要顯示的每個圖形建立一個GraphSet,並將圖形資料新增到其中。
為圖表上的每個軌道建立一個Track,並將GraphSets和FeatureSets新增到所需的軌道中。
建立一個Diagram,並將Tracks新增到其中。
告訴Diagram繪製圖像。
將影像寫入檔案。
讓我們以一個輸入GenBank檔案為例:
https://raw.githubusercontent.com/biopython/biopython/master/Doc/examples/ls_orchid.gbk 並從SeqRecord物件中讀取記錄,然後最終繪製基因組圖。解釋如下:
我們將首先匯入所有模組,如下所示:
>>> from reportlab.lib import colors >>> from reportlab.lib.units import cm >>> from Bio.Graphics import GenomeDiagram
現在,匯入SeqIO模組以讀取資料:
>>> from Bio import SeqIO
record = SeqIO.read("example.gb", "genbank")
這裡,record從genbank檔案中讀取序列。
現在,建立一個空圖表以新增軌道和特徵集:
>>> diagram = GenomeDiagram.Diagram( "Yersinia pestis biovar Microtus plasmid pPCP1") >>> track = diagram.new_track(1, name="Annotated Features") >>> feature = track.new_set()
現在,我們可以使用從綠色到灰色的備用顏色(如下定義)應用顏色主題更改:
>>> for feature in record.features: >>> if feature.type != "gene": >>> continue >>> if len(feature) % 2 == 0: >>> color = colors.blue >>> else: >>> color = colors.red >>> >>> feature.add_feature(feature, color=color, label=True)
現在您可以在螢幕上看到以下響應:
<Bio.Graphics.GenomeDiagram._Feature.Feature object at 0x105d3dc90> <Bio.Graphics.GenomeDiagram._Feature.Feature object at 0x105d3dfd0> <Bio.Graphics.GenomeDiagram._Feature.Feature object at 0x1007627d0> <Bio.Graphics.GenomeDiagram._Feature.Feature object at 0x105d57290> <Bio.Graphics.GenomeDiagram._Feature.Feature object at 0x105d57050> <Bio.Graphics.GenomeDiagram._Feature.Feature object at 0x105d57390> <Bio.Graphics.GenomeDiagram._Feature.Feature object at 0x105d57590> <Bio.Graphics.GenomeDiagram._Feature.Feature object at 0x105d57410> <Bio.Graphics.GenomeDiagram._Feature.Feature object at 0x105d57490> <Bio.Graphics.GenomeDiagram._Feature.Feature object at 0x105d574d0>
讓我們為上述輸入記錄繪製一個圖:
>>> diagram.draw(
format = "linear", orientation = "landscape", pagesize = 'A4',
... fragments = 4, start = 0, end = len(record))
>>> diagram.write("orchid.pdf", "PDF")
>>> diagram.write("orchid.eps", "EPS")
>>> diagram.write("orchid.svg", "SVG")
>>> diagram.write("orchid.png", "PNG")
執行上述命令後,您可以在Biopython目錄中看到儲存的以下影像。
** Result ** genome.png
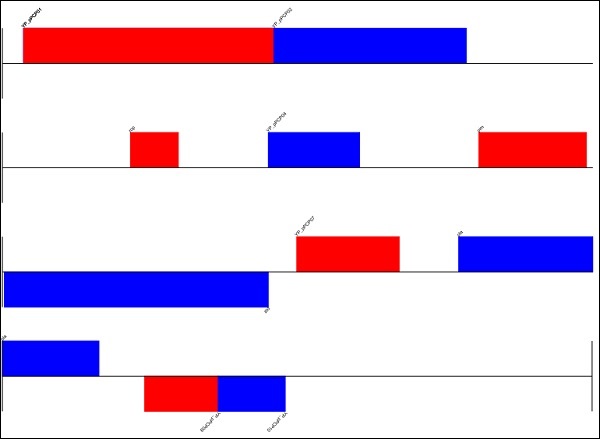
您還可以透過進行以下更改以圓形格式繪製圖像:
>>> diagram.draw(
format = "circular", circular = True, pagesize = (20*cm,20*cm),
... start = 0, end = len(record), circle_core = 0.7)
>>> diagram.write("circular.pdf", "PDF")
染色體概述
DNA分子被包裝成稱為染色體的線狀結構。每條染色體都由DNA組成,DNA緊密纏繞在稱為組蛋白的蛋白質周圍多次,這些蛋白質支撐著它的結構。
當細胞不分裂時,染色體在細胞核中不可見——甚至在顯微鏡下也看不到。但是,構成染色體的DNA在細胞分裂過程中會變得更加緊密地包裝,然後可以在顯微鏡下看到。
在人類中,每個細胞通常包含23對染色體,總共46條。其中22對稱為常染色體,在男性和女性中看起來相同。第23對,性染色體,在男性和女性之間有所不同。女性有兩條X染色體的複製,而男性有一條X染色體和一條Y染色體。
Biopython - 表型微陣列
表型被定義為生物體針對特定化學物質或環境表現出的可觀察特徵或性狀。表型微陣列同時測量生物體對大量化學物質和環境的反應,並分析資料以瞭解基因突變、基因特徵等。
Biopython提供了一個優秀的模組Bio.Phenotype來分析表型資料。讓我們在本節中學習如何解析、內插、提取和分析表型微陣列資料。
解析
表型微陣列資料可以採用兩種格式:CSV和JSON。Biopython支援這兩種格式。Biopython解析器解析表型微陣列資料並將其作為PlateRecord物件的集合返回。每個PlateRecord物件包含WellRecord物件的集合。每個WellRecord物件以8行12列的格式儲存資料。八行由A到H表示,十二列由01到12表示。例如,第4行和第6列由D06表示。
讓我們透過以下示例瞭解格式和解析的概念:
步驟1 - 下載Biopython團隊提供的Plates.csv檔案:https://raw.githubusercontent.com/biopython/biopython/master/Doc/examples/Plates.csv
步驟2 - 如下載入phenotpe模組:
>>> from Bio import phenotype
步驟3 - 呼叫phenotype.parse方法,傳遞資料檔案和格式選項(“pm-csv”)。它返回可迭代的PlateRecord,如下所示:
>>> plates = list(phenotype.parse('Plates.csv', "pm-csv"))
>>> plates
[PlateRecord('WellRecord['A01'], WellRecord['A02'], WellRecord['A03'], ..., WellRecord['H12']'),
PlateRecord('WellRecord['A01'], WellRecord['A02'], WellRecord['A03'], ..., WellRecord['H12']'),
PlateRecord('WellRecord['A01'], WellRecord['A02'], WellRecord['A03'], ..., WellRecord['H12']'),
PlateRecord('WellRecord['A01'], WellRecord['A02'],WellRecord['A03'], ..., WellRecord['H12']')]
>>>
步驟4 - 從列表中訪問第一個板,如下所示:
>>> plate = plates[0]
>>> plate
PlateRecord('WellRecord['A01'], WellRecord['A02'], WellRecord['A03'], ...,
WellRecord['H12']')
>>>
步驟5 - 如前所述,一個板包含8行,每行有12個專案。可以以兩種方式訪問WellRecord,如下所示:
>>> well = plate["A04"]
>>> well = plate[0, 4]
>>> well WellRecord('(0.0, 0.0), (0.25, 0.0), (0.5, 0.0), (0.75, 0.0),
(1.0, 0.0), ..., (71.75, 388.0)')
>>>
步驟6 - 每個孔將在不同的時間點具有一系列測量值,並且可以使用for迴圈訪問,如下所示:
>>> for v1, v2 in well: ... print(v1, v2) ... 0.0 0.0 0.25 0.0 0.5 0.0 0.75 0.0 1.0 0.0 ... 71.25 388.0 71.5 388.0 71.75 388.0 >>>
插值
插值可以更深入地瞭解資料。Biopython提供方法來插值WellRecord資料,以獲取中間時間點的資訊。語法類似於列表索引,因此易於學習。
要獲取20.1小時的資料,只需將索引值傳遞如下所示:
>>> well[20.10] 69.40000000000003 >>>
我們也可以傳遞開始時間點和結束時間點,如下所示:
>>> well[20:30] [67.0, 84.0, 102.0, 119.0, 135.0, 147.0, 158.0, 168.0, 179.0, 186.0] >>>
以上命令將資料從 20 小時插值到 30 小時,時間間隔為 1 小時。預設情況下,時間間隔為 1 小時,我們可以將其更改為任何值。例如,讓我們指定如下所示的 15 分鐘(0.25 小時)間隔:
>>> well[20:21:0.25] [67.0, 73.0, 75.0, 81.0] >>>
分析和提取
Biopython 提供了一種 fit 方法,可以使用 Gompertz、Logistic 和 Richards S 型函式分析 WellRecord 資料。預設情況下,fit 方法使用 Gompertz 函式。我們需要呼叫 WellRecord 物件的 fit 方法來完成此任務。程式碼如下:
>>> well.fit() Traceback (most recent call last): ... Bio.MissingPythonDependencyError: Install scipy to extract curve parameters. >>> well.model >>> getattr(well, 'min') 0.0 >>> getattr(well, 'max') 388.0 >>> getattr(well, 'average_height') 205.42708333333334 >>>
Biopython 依賴於 scipy 模組進行高階分析。它將計算最小值、最大值和平均高度詳細資訊,而無需使用 scipy 模組。
Biopython - 繪圖
本章介紹如何繪製序列圖。在進入這個主題之前,讓我們先了解繪圖的基本知識。
繪圖
Matplotlib 是一個 Python 繪相簿,它可以生成各種格式的高質量圖形。我們可以建立不同型別的繪圖,例如折線圖、直方圖、條形圖、餅圖、散點圖等。
pyLab 是屬於 matplotlib 的一個模組,它將數值模組 numpy 與圖形繪圖模組 pyplot 相結合。Biopython 使用 pylab 模組繪製序列圖。為此,我們需要匯入以下程式碼:
import pylab
在匯入之前,我們需要使用以下命令使用 pip 命令安裝 matplotlib 包:
pip install matplotlib
示例輸入檔案
在您的 Biopython 目錄中建立一個名為 plot.fasta 的示例檔案,並新增以下更改:
>seq0 FQTWEEFSRAAEKLYLADPMKVRVVLKYRHVDGNLCIKVTDDLVCLVYRTDQAQDVKKIEKF >seq1 KYRTWEEFTRAAEKLYQADPMKVRVVLKYRHCDGNLCIKVTDDVVCLLYRTDQAQDVKKIEKFHSQLMRLME >seq2 EEYQTWEEFARAAEKLYLTDPMKVRVVLKYRHCDGNLCMKVTDDAVCLQYKTDQAQDVKKVEKLHGK >seq3 MYQVWEEFSRAVEKLYLTDPMKVRVVLKYRHCDGNLCIKVTDNSVCLQYKTDQAQDV >seq4 EEFSRAVEKLYLTDPMKVRVVLKYRHCDGNLCIKVTDNSVVSYEMRLFGVQKDNFALEHSLL >seq5 SWEEFAKAAEVLYLEDPMKCRMCTKYRHVDHKLVVKLTDNHTVLKYVTDMAQDVKKIEKLTTLLMR >seq6 FTNWEEFAKAAERLHSANPEKCRFVTKYNHTKGELVLKLTDDVVCLQYSTNQLQDVKKLEKLSSTLLRSI >seq7 SWEEFVERSVQLFRGDPNATRYVMKYRHCEGKLVLKVTDDRECLKFKTDQAQDAKKMEKLNNIFF >seq8 SWDEFVDRSVQLFRADPESTRYVMKYRHCDGKLVLKVTDNKECLKFKTDQAQEAKKMEKLNNIFFTLM >seq9 KNWEDFEIAAENMYMANPQNCRYTMKYVHSKGHILLKMSDNVKCVQYRAENMPDLKK >seq10 FDSWDEFVSKSVELFRNHPDTTRYVVKYRHCEGKLVLKVTDNHECLKFKTDQAQDAKKMEK
折線圖
現在,讓我們為上述 fasta 檔案建立一個簡單的折線圖。
步驟 1 - 匯入 SeqIO 模組以讀取 fasta 檔案。
>>> from Bio import SeqIO
步驟 2 - 解析輸入檔案。
>>> records = [len(rec) for rec in SeqIO.parse("plot.fasta", "fasta")]
>>> len(records)
11
>>> max(records)
72
>>> min(records)
57
步驟 3 - 讓我們匯入 pylab 模組。
>>> import pylab
步驟 4 - 透過分配 x 軸和 y 軸標籤來配置折線圖。
>>> pylab.xlabel("sequence length")
Text(0.5, 0, 'sequence length')
>>> pylab.ylabel("count")
Text(0, 0.5, 'count')
>>>
步驟 5 - 透過設定網格顯示來配置折線圖。
>>> pylab.grid()
步驟 6 - 透過呼叫 plot 方法並將記錄作為輸入來繪製簡單的折線圖。
>>> pylab.plot(records) [<matplotlib.lines.Line2D object at 0x10b6869d 0>]
步驟 7 - 最後使用以下命令儲存圖表。
>>> pylab.savefig("lines.png")
結果
執行上述命令後,您可以在Biopython目錄中看到儲存的以下影像。
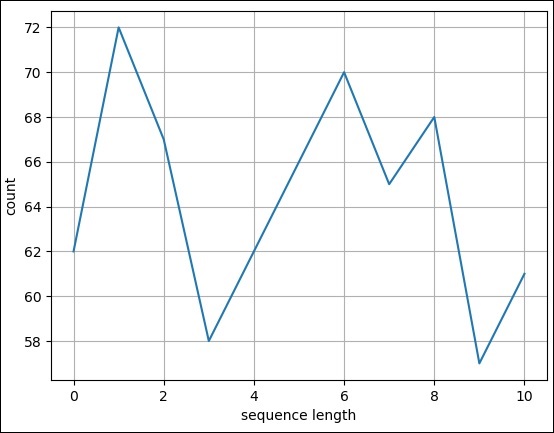
直方圖
直方圖用於連續資料,其中 bin 表示資料範圍。繪製直方圖與折線圖相同,只是使用 pylab.hist 而不是 pylab.plot。改為呼叫 pylab 模組的 hist 方法,並使用記錄和一些自定義 bin 值(5)。完整的程式碼如下:
步驟 1 - 匯入 SeqIO 模組以讀取 fasta 檔案。
>>> from Bio import SeqIO
步驟 2 - 解析輸入檔案。
>>> records = [len(rec) for rec in SeqIO.parse("plot.fasta", "fasta")]
>>> len(records)
11
>>> max(records)
72
>>> min(records)
57
步驟 3 - 讓我們匯入 pylab 模組。
>>> import pylab
步驟 4 - 透過分配 x 軸和 y 軸標籤來配置折線圖。
>>> pylab.xlabel("sequence length")
Text(0.5, 0, 'sequence length')
>>> pylab.ylabel("count")
Text(0, 0.5, 'count')
>>>
步驟 5 - 透過設定網格顯示來配置折線圖。
>>> pylab.grid()
步驟 6 - 透過呼叫 plot 方法並將記錄作為輸入來繪製簡單的折線圖。
>>> pylab.hist(records,bins=5) (array([2., 3., 1., 3., 2.]), array([57., 60., 63., 66., 69., 72.]), <a list of 5 Patch objects>) >>>
步驟 7 - 最後使用以下命令儲存圖表。
>>> pylab.savefig("hist.png")
結果
執行上述命令後,您可以在Biopython目錄中看到儲存的以下影像。
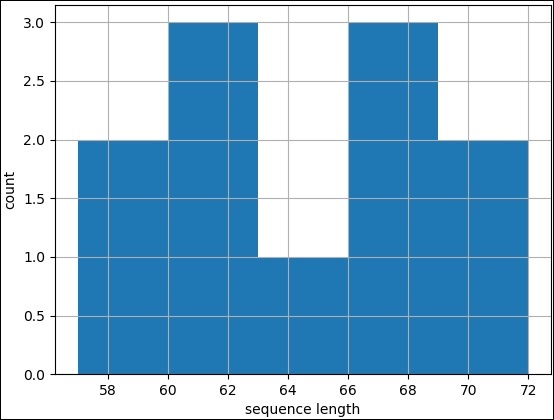
序列中的 GC 百分比
GC 百分比是用於比較不同序列的常用分析資料之一。我們可以使用一組序列的 GC 百分比建立一個簡單的折線圖,並立即進行比較。在這裡,我們可以將資料從序列長度更改為 GC 百分比。完整的程式碼如下:
步驟 1 - 匯入 SeqIO 模組以讀取 fasta 檔案。
>>> from Bio import SeqIO
步驟 2 - 解析輸入檔案。
>>> from Bio.SeqUtils import GC
>>> gc = sorted(GC(rec.seq) for rec in SeqIO.parse("plot.fasta", "fasta"))
步驟 3 - 讓我們匯入 pylab 模組。
>>> import pylab
步驟 4 - 透過分配 x 軸和 y 軸標籤來配置折線圖。
>>> pylab.xlabel("Genes")
Text(0.5, 0, 'Genes')
>>> pylab.ylabel("GC Percentage")
Text(0, 0.5, 'GC Percentage')
>>>
步驟 5 - 透過設定網格顯示來配置折線圖。
>>> pylab.grid()
步驟 6 - 透過呼叫 plot 方法並將記錄作為輸入來繪製簡單的折線圖。
>>> pylab.plot(gc) [<matplotlib.lines.Line2D object at 0x10b6869d 0>]
步驟 7 - 最後使用以下命令儲存圖表。
>>> pylab.savefig("gc.png")
結果
執行上述命令後,您可以在Biopython目錄中看到儲存的以下影像。
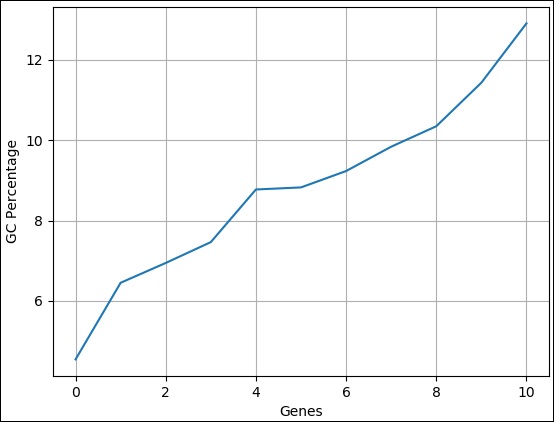
Biopython - 聚類分析
通常,聚類分析是將一組物件分組到同一組中。這個概念主要用於資料探勘、統計資料分析、機器學習、模式識別、影像分析、生物資訊學等。它可以透過各種演算法來實現,以瞭解聚類在不同分析中的廣泛應用。
根據生物資訊學,聚類分析主要用於基因表達資料分析,以查詢具有相似基因表達的基因組。
在本章中,我們將檢查 Biopython 中重要的演算法,以瞭解在真實資料集上進行聚類的基礎知識。
Biopython 使用 Bio.Cluster 模組來實現所有演算法。它支援以下演算法:
- 層次聚類
- K 均值聚類
- 自組織對映
- 主成分分析
讓我們簡要介紹一下上述演算法。
層次聚類
層次聚類用於透過距離度量將每個節點與其最近的鄰居連線起來並建立一個聚類。Bio.Cluster 節點具有三個屬性:left、right 和 distance。讓我們建立一個簡單的聚類,如下所示:
>>> from Bio.Cluster import Node >>> n = Node(1,10) >>> n.left = 11 >>> n.right = 0 >>> n.distance = 1 >>> print(n) (11, 0): 1
如果要構建基於樹的聚類,請使用以下命令:
>>> n1 = [Node(1, 2, 0.2), Node(0, -1, 0.5)] >>> n1_tree = Tree(n1) >>> print(n1_tree) (1, 2): 0.2 (0, -1): 0.5 >>> print(n1_tree[0]) (1, 2): 0.2
讓我們使用 Bio.Cluster 模組執行層次聚類。
假設距離是在一個數組中定義的。
>>> import numpy as np >>> distance = array([[1,2,3],[4,5,6],[3,5,7]])
現在將距離陣列新增到樹狀聚類中。
>>> from Bio.Cluster import treecluster >>> cluster = treecluster(distance) >>> print(cluster) (2, 1): 0.666667 (-1, 0): 9.66667
上述函式返回一個 Tree 聚類物件。此物件包含節點,其中專案數被聚類為行或列。
K 均值聚類
這是一種分割槽演算法,分為 k 均值、中位數和中心點聚類。讓我們簡要了解每種聚類。
K 均值聚類
這種方法在資料探勘中很流行。該演算法的目標是在資料中查詢組,其中組數由變數 K 表示。
該演算法迭代地將每個資料點分配到 K 個組中的一個,這基於提供的特徵。資料點根據特徵相似性進行聚類。
>>> from Bio.Cluster import kcluster >>> from numpy import array >>> data = array([[1, 2], [3, 4], [5, 6]]) >>> clusterid, error,found = kcluster(data) >>> print(clusterid) [0 0 1] >>> print(found) 1
K 中位數聚類
這是另一種聚類演算法,它計算每個聚類的均值以確定其質心。
K 中心點聚類
這種方法基於給定的一組專案,使用使用者傳遞的距離矩陣和聚類數。
假設距離矩陣定義如下:
>>> distance = array([[1,2,3],[4,5,6],[3,5,7]])
我們可以使用以下命令計算 k 中心點聚類:
>>> from Bio.Cluster import kmedoids >>> clusterid, error, found = kmedoids(distance)
讓我們考慮一個例子。
kcluster 函式將資料矩陣作為輸入,而不是 Seq 例項。您需要將序列轉換為矩陣並將其提供給 kcluster 函式。
將資料轉換為僅包含數值元素的矩陣的一種方法是使用 numpy.fromstring 函式。它基本上將序列中的每個字母轉換為其 ASCII 對應項。
這將建立一個編碼序列的二維陣列,kcluster 函式識別該陣列並使用它來對序列進行聚類。
>>> from Bio.Cluster import kcluster >>> import numpy as np >>> sequence = [ 'AGCT','CGTA','AAGT','TCCG'] >>> matrix = np.asarray([np.fromstring(s, dtype=np.uint8) for s in sequence]) >>> clusterid,error,found = kcluster(matrix) >>> print(clusterid) [1 0 0 1]
自組織對映
這種方法是一種人工神經網路。它由 Kohonen 開發,通常稱為 Kohonen 對映。它根據矩形拓撲將專案組織成聚類。
讓我們使用與以下相同的陣列距離建立一個簡單的聚類:
>>> from Bio.Cluster import somcluster >>> from numpy import array >>> data = array([[1, 2], [3, 4], [5, 6]]) >>> clusterid,map = somcluster(data) >>> print(map) [[[-1.36032469 0.38667395]] [[-0.41170578 1.35295911]]] >>> print(clusterid) [[1 0] [1 0] [1 0]]
這裡,clusterid 是一個有兩列的陣列,其中行數等於聚類的專案數,而 data 是一個維度為行或列的陣列。
主成分分析
主成分分析可用於視覺化高維資料。這是一種使用來自線性代數和統計學的簡單矩陣運算來計算原始資料投影到相同數量或更少維度的方法。
主成分分析返回一個元組 columnmean、coordinates、components 和 eigenvalues。讓我們瞭解一下這個概念的基本知識。
>>> from numpy import array >>> from numpy import mean >>> from numpy import cov >>> from numpy.linalg import eig # define a matrix >>> A = array([[1, 2], [3, 4], [5, 6]]) >>> print(A) [[1 2] [3 4] [5 6]] # calculate the mean of each column >>> M = mean(A.T, axis = 1) >>> print(M) [ 3. 4.] # center columns by subtracting column means >>> C = A - M >>> print(C) [[-2. -2.] [ 0. 0.] [ 2. 2.]] # calculate covariance matrix of centered matrix >>> V = cov(C.T) >>> print(V) [[ 4. 4.] [ 4. 4.]] # eigendecomposition of covariance matrix >>> values, vectors = eig(V) >>> print(vectors) [[ 0.70710678 -0.70710678] [ 0.70710678 0.70710678]] >>> print(values) [ 8. 0.]
讓我們將相同的矩形矩陣資料應用於 Bio.Cluster 模組,如下所示:
>>> from Bio.Cluster import pca >>> from numpy import array >>> data = array([[1, 2], [3, 4], [5, 6]]) >>> columnmean, coordinates, components, eigenvalues = pca(data) >>> print(columnmean) [ 3. 4.] >>> print(coordinates) [[-2.82842712 0. ] [ 0. 0. ] [ 2.82842712 0. ]] >>> print(components) [[ 0.70710678 0.70710678] [ 0.70710678 -0.70710678]] >>> print(eigenvalues) [ 4. 0.]
Biopython - 機器學習
生物資訊學是應用機器學習演算法的極佳領域。在這裡,我們擁有大量生物體的遺傳資訊,不可能手動分析所有這些資訊。如果使用合適的機器學習演算法,我們可以從這些資料中提取大量有用的資訊。Biopython 提供了一套有用的演算法來進行監督式機器學習。
監督學習基於輸入變數 (X) 和輸出變數 (Y)。它使用一種演算法來學習從輸入到輸出的對映函式。它定義如下:
Y = f(X)
這種方法的主要目標是近似對映函式,當您有新的輸入資料 (x) 時,您可以預測該資料的輸出變數 (Y)。
邏輯迴歸模型
邏輯迴歸是一種監督式機器學習演算法。它用於使用預測變數的加權和找出 K 個類別之間的差異。它計算事件發生的機率,可用於癌症檢測。
Biopython 提供 Bio.LogisticRegression 模組來根據邏輯迴歸演算法預測變數。目前,Biopython 僅實現了用於兩個類別的邏輯迴歸演算法 (K = 2)。
K 近鄰
K 近鄰也是一種監督式機器學習演算法。它透過根據最近鄰對資料進行分類來工作。Biopython 提供 Bio.KNN 模組來根據 k 近鄰演算法預測變數。
樸素貝葉斯
樸素貝葉斯分類器是一組基於貝葉斯定理的分類演算法。它不是單個演算法,而是一系列演算法,它們都共享一個共同的原則,即要分類的每對特徵彼此獨立。Biopython 提供 Bio.NaiveBayes 模組來使用樸素貝葉斯演算法。
馬爾可夫模型
馬爾可夫模型是一個數學系統,定義為一組隨機變數,根據某些機率規則經歷從一個狀態到另一個狀態的轉換。Biopython 提供 Bio.MarkovModel 和 Bio.HMM.MarkovModel 模組來使用馬爾可夫模型。
Biopython - 測試技術
Biopython 擁有廣泛的測試指令碼,用於在不同條件下測試軟體,以確保軟體沒有錯誤。要執行測試指令碼,請下載 Biopython 的原始碼,然後執行以下命令:
python run_tests.py
這將執行所有測試指令碼並給出以下輸出:
Python version: 2.7.12 (v2.7.12:d33e0cf91556, Jun 26 2016, 12:10:39) [GCC 4.2.1 (Apple Inc. build 5666) (dot 3)] Operating system: posix darwin test_Ace ... ok test_Affy ... ok test_AlignIO ... ok test_AlignIO_ClustalIO ... ok test_AlignIO_EmbossIO ... ok test_AlignIO_FastaIO ... ok test_AlignIO_MauveIO ... ok test_AlignIO_PhylipIO ... ok test_AlignIO_convert ... ok ........................................... ...........................................
我們還可以執行指定的單個測試指令碼,如下所示:
python test_AlignIO.py
結論
正如我們所學到的,Biopython 是生物資訊學領域的重要軟體之一。它用 Python 編寫(易於學習和編寫),提供了廣泛的功能來處理生物資訊學領域中的任何計算和操作。它還為幾乎所有流行的生物資訊學軟體提供了簡單靈活的介面,以利用其功能。