- Biopython 教程
- Biopython - 首頁
- Biopython - 簡介
- Biopython - 安裝
- 建立簡單應用程式
- Biopython - 序列
- 高階序列操作
- 序列 I/O 操作
- Biopython - 序列比對
- Biopython - BLAST 概述
- Biopython - Entrez 資料庫
- Biopython - PDB 模組
- Biopython - 基序物件
- Biopython - BioSQL 模組
- Biopython - 種群遺傳學
- Biopython - 基因組分析
- Biopython - 表型微陣列
- Biopython - 繪圖
- Biopython - 聚類分析
- Biopython - 機器學習
- Biopython - 測試技術
- Biopython 資源
- Biopython - 快速指南
- Biopython - 有用資源
- Biopython - 討論
Biopython - 繪圖
本章介紹如何繪製序列圖。在進入這個主題之前,讓我們先了解繪製圖的基本知識。
繪圖
Matplotlib 是一個 Python 繪相簿,它可以生成各種格式的高質量圖形。我們可以建立不同型別的繪圖,例如折線圖、直方圖、條形圖、餅圖、散點圖等。
pyLab 是 matplotlib 中的一個模組,它將數值模組 numpy 與圖形繪製模組 pyplot 結合在一起。Biopython 使用 pylab 模組繪製序列圖。為此,我們需要匯入以下程式碼:
import pylab
在匯入之前,我們需要使用 pip 命令安裝 matplotlib 包,命令如下:
pip install matplotlib
示例輸入檔案
在您的 Biopython 目錄中建立一個名為 plot.fasta 的示例檔案,並新增以下內容:
>seq0 FQTWEEFSRAAEKLYLADPMKVRVVLKYRHVDGNLCIKVTDDLVCLVYRTDQAQDVKKIEKF >seq1 KYRTWEEFTRAAEKLYQADPMKVRVVLKYRHCDGNLCIKVTDDVVCLLYRTDQAQDVKKIEKFHSQLMRLME >seq2 EEYQTWEEFARAAEKLYLTDPMKVRVVLKYRHCDGNLCMKVTDDAVCLQYKTDQAQDVKKVEKLHGK >seq3 MYQVWEEFSRAVEKLYLTDPMKVRVVLKYRHCDGNLCIKVTDNSVCLQYKTDQAQDV >seq4 EEFSRAVEKLYLTDPMKVRVVLKYRHCDGNLCIKVTDNSVVSYEMRLFGVQKDNFALEHSLL >seq5 SWEEFAKAAEVLYLEDPMKCRMCTKYRHVDHKLVVKLTDNHTVLKYVTDMAQDVKKIEKLTTLLMR >seq6 FTNWEEFAKAAERLHSANPEKCRFVTKYNHTKGELVLKLTDDVVCLQYSTNQLQDVKKLEKLSSTLLRSI >seq7 SWEEFVERSVQLFRGDPNATRYVMKYRHCEGKLVLKVTDDRECLKFKTDQAQDAKKMEKLNNIFF >seq8 SWDEFVDRSVQLFRADPESTRYVMKYRHCDGKLVLKVTDNKECLKFKTDQAQEAKKMEKLNNIFFTLM >seq9 KNWEDFEIAAENMYMANPQNCRYTMKYVHSKGHILLKMSDNVKCVQYRAENMPDLKK >seq10 FDSWDEFVSKSVELFRNHPDTTRYVVKYRHCEGKLVLKVTDNHECLKFKTDQAQDAKKMEK
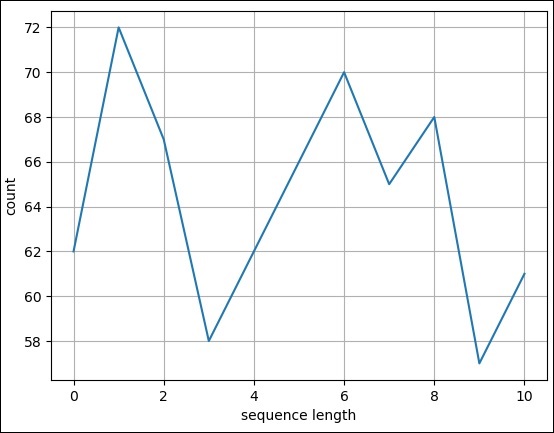
折線圖
現在,讓我們為上述 fasta 檔案建立一個簡單的折線圖。
步驟 1 - 匯入 SeqIO 模組以讀取 fasta 檔案。
>>> from Bio import SeqIO
步驟 2 - 解析輸入檔案。
>>> records = [len(rec) for rec in SeqIO.parse("plot.fasta", "fasta")]
>>> len(records)
11
>>> max(records)
72
>>> min(records)
57
步驟 3 - 讓我們匯入 pylab 模組。
>>> import pylab
步驟 4 - 透過分配 x 軸和 y 軸標籤來配置折線圖。
>>> pylab.xlabel("sequence length")
Text(0.5, 0, 'sequence length')
>>> pylab.ylabel("count")
Text(0, 0.5, 'count')
>>>
步驟 5 - 透過設定網格顯示來配置折線圖。
>>> pylab.grid()
步驟 6 - 透過呼叫 plot 方法並提供記錄作為輸入來繪製簡單的折線圖。
>>> pylab.plot(records) [<matplotlib.lines.Line2D object at 0x10b6869d 0>]
步驟 7 - 最後,使用以下命令儲存圖表。
>>> pylab.savefig("lines.png")
結果
執行上述命令後,您可以在 Biopython 目錄中看到儲存的以下影像。
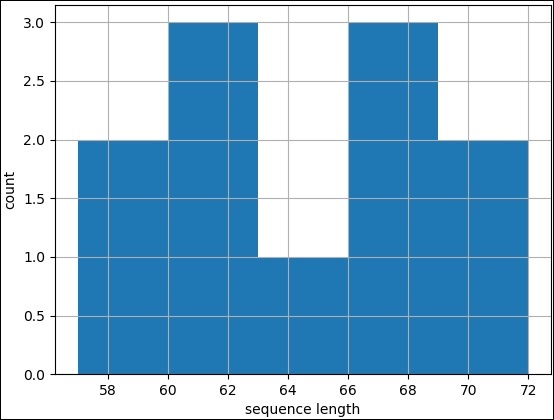
直方圖
直方圖用於連續資料,其中 bin 表示資料範圍。繪製直方圖與折線圖相同,只是不使用 pylab.plot。而是使用 pylab 模組的 hist 方法,並使用記錄和一些自定義的 bin 值(5)。完整的程式碼如下:
步驟 1 - 匯入 SeqIO 模組以讀取 fasta 檔案。
>>> from Bio import SeqIO
步驟 2 - 解析輸入檔案。
>>> records = [len(rec) for rec in SeqIO.parse("plot.fasta", "fasta")]
>>> len(records)
11
>>> max(records)
72
>>> min(records)
57
步驟 3 - 讓我們匯入 pylab 模組。
>>> import pylab
步驟 4 - 透過分配 x 軸和 y 軸標籤來配置折線圖。
>>> pylab.xlabel("sequence length")
Text(0.5, 0, 'sequence length')
>>> pylab.ylabel("count")
Text(0, 0.5, 'count')
>>>
步驟 5 - 透過設定網格顯示來配置折線圖。
>>> pylab.grid()
步驟 6 - 透過呼叫 plot 方法並提供記錄作為輸入來繪製簡單的折線圖。
>>> pylab.hist(records,bins=5) (array([2., 3., 1., 3., 2.]), array([57., 60., 63., 66., 69., 72.]), <a list of 5 Patch objects>) >>>
步驟 7 - 最後,使用以下命令儲存圖表。
>>> pylab.savefig("hist.png")
結果
執行上述命令後,您可以在 Biopython 目錄中看到儲存的以下影像。
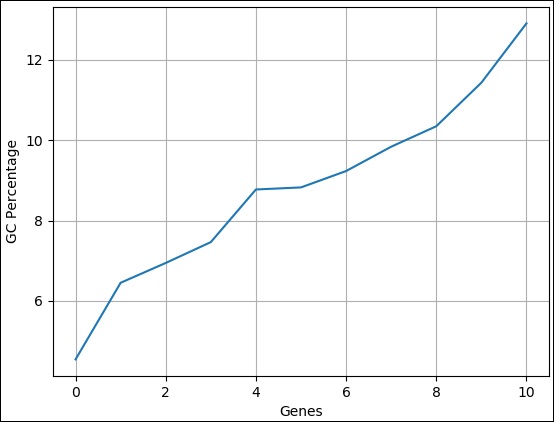
序列中的 GC 百分比
GC 百分比是用於比較不同序列的常用分析資料之一。我們可以使用一組序列的 GC 百分比建立一個簡單的折線圖,並立即進行比較。在這裡,我們只需將資料從序列長度更改為 GC 百分比。完整的程式碼如下:
步驟 1 - 匯入 SeqIO 模組以讀取 fasta 檔案。
>>> from Bio import SeqIO
步驟 2 - 解析輸入檔案。
>>> from Bio.SeqUtils import GC
>>> gc = sorted(GC(rec.seq) for rec in SeqIO.parse("plot.fasta", "fasta"))
步驟 3 - 讓我們匯入 pylab 模組。
>>> import pylab
步驟 4 - 透過分配 x 軸和 y 軸標籤來配置折線圖。
>>> pylab.xlabel("Genes")
Text(0.5, 0, 'Genes')
>>> pylab.ylabel("GC Percentage")
Text(0, 0.5, 'GC Percentage')
>>>
步驟 5 - 透過設定網格顯示來配置折線圖。
>>> pylab.grid()
步驟 6 - 透過呼叫 plot 方法並提供記錄作為輸入來繪製簡單的折線圖。
>>> pylab.plot(gc) [<matplotlib.lines.Line2D object at 0x10b6869d 0>]
步驟 7 - 最後,使用以下命令儲存圖表。
>>> pylab.savefig("gc.png")
結果
執行上述命令後,您可以在 Biopython 目錄中看到儲存的以下影像。