在 Matplotlib 中繪製垂直線
Python 提供了一個名為 Matplotlib 的強大庫,該庫以繪圖和圖形的形式建立視覺化表示。該庫的眾多功能之一是能夠繪製垂直線,這些垂直線用於新增參考線或突出顯示給定繪圖上的特定點。Matplotlib 的內建方法 'axvline()'、'vlines()' 和 'plot()' 用於繪製垂直線,並具有可自定義的引數,例如位置、顏色和線型。我們將藉助示例程式來解釋這些方法。
使用 Matplotlib 繪製垂直線
讓我們討論 Matplotlib 的內建方法及其示例程式,這些程式將向我們展示如何繪製垂直線。
使用 axvline() 方法
這是在 Matplotlib 中繪製垂直線的最簡單方法。使用此方法的優點是簡單方便。但是,缺點是它不允許我們指定線的 y 範圍。axvline() 方法接受一個引數,該引數指定垂直線的 x 座標。它可以接受稍後我們將討論的其他引數。
語法
axvline(x = position)
此處,position 指定垂直線在 x 座標上的位置。
示例 1
在此示例中,我們將繪製一條跨越繪圖整個 y 軸的垂直線。該線在 x 座標上的位置將為 2。
import matplotlib.pyplot as plt plt.axvline(x = 2) plt.show()
輸出
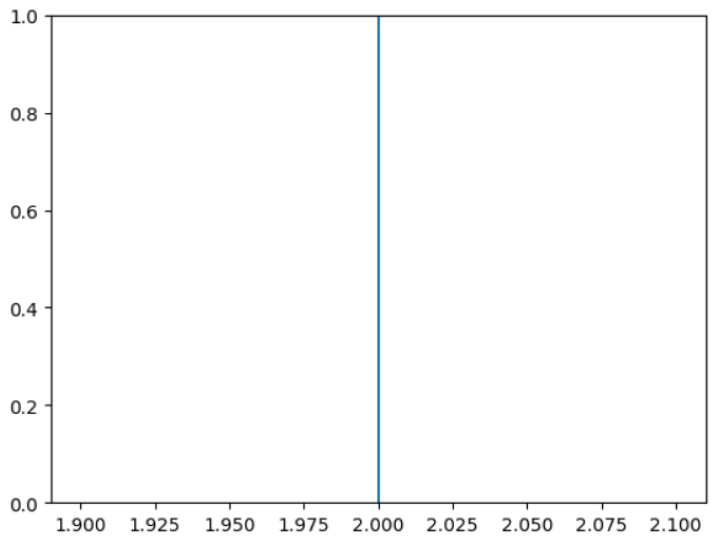
示例 2
這是另一個繪製跨越繪圖整個 y 軸的垂直線的示例。但是這次,我們將透過向 axvline() 傳遞其他引數(例如顏色、線型和 alpha)來自定義線的外觀。
import matplotlib.pyplot as plt plt.axvline(x = 2, color = 'red', linestyle = '--', alpha = 0.5) plt.show()
輸出
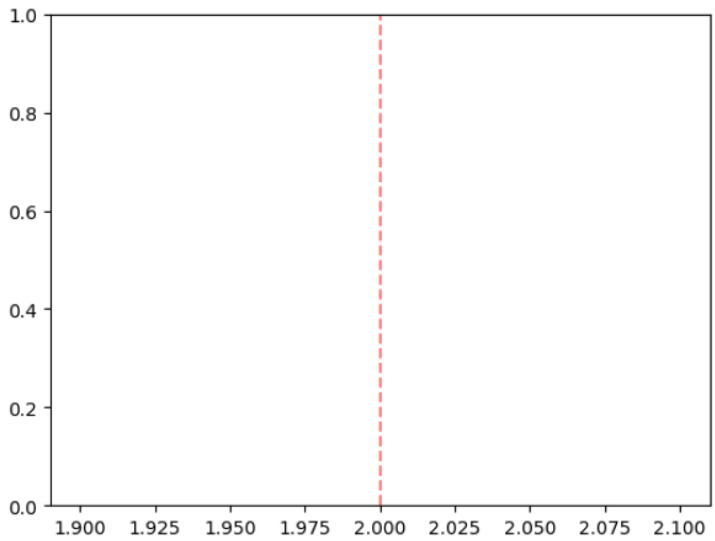
我們在 x 座標上的位置 2 繪製了一條虛線紅色線,透明度為 50%。
使用 plot() 方法
這是在 Matplotlib 中繪製垂直線的另一種方法。與 axvline() 不同,它允許我們指定線的 y 範圍。要使用 plot() 繪製垂直線,請將具有相同 x 座標和不同 y 座標的兩個點作為引數傳遞。
語法
plot([x1, x2], [y1, y2])
示例 1
在下面的示例中,我們將繪製一條在 x 座標上的位置 2 以及在 y 座標上的 1 到 3 之間繪製的垂直線。
import matplotlib.pyplot as plt plt.plot([2, 2], [1, 3]) plt.show()
輸出
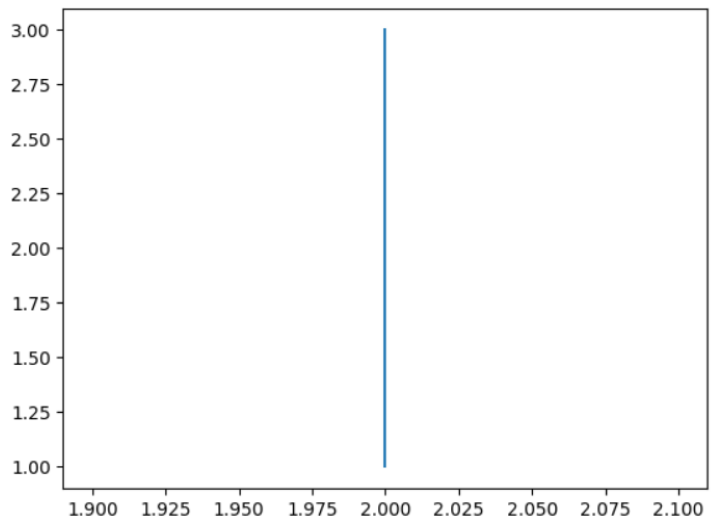
示例 2
在此示例中,我們將透過向 plot() 傳遞其他引數(例如顏色、線型和標記)來自定義線的外觀。
import matplotlib.pyplot as plt plt.plot([2, 2], [1, 3], color = 'blue', linestyle = ':', marker = 'o') plt.show()
輸出
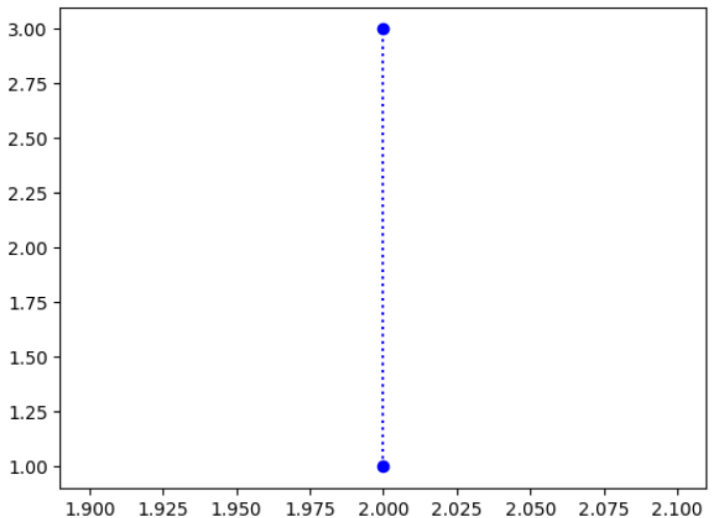
在上面的程式碼中,我們繪製了一條帶有圓圈端點的點狀藍色線。
使用 vlines() 方法
這是在 Matplotlib 中繪製垂直線的第三種方法。它接受三個引數:x、ymin 和 ymax,它們分別是垂直線的 x 座標以及下限和上限 y 座標。
語法
vlines(x = position, ymin = position, ymax = position)
示例 1
以下示例說明了如何使用 vlines() 繪製垂直線。
import matplotlib.pyplot as plt plt.vlines(x = 3, ymin = 1, ymax = 3) plt.show()
輸出
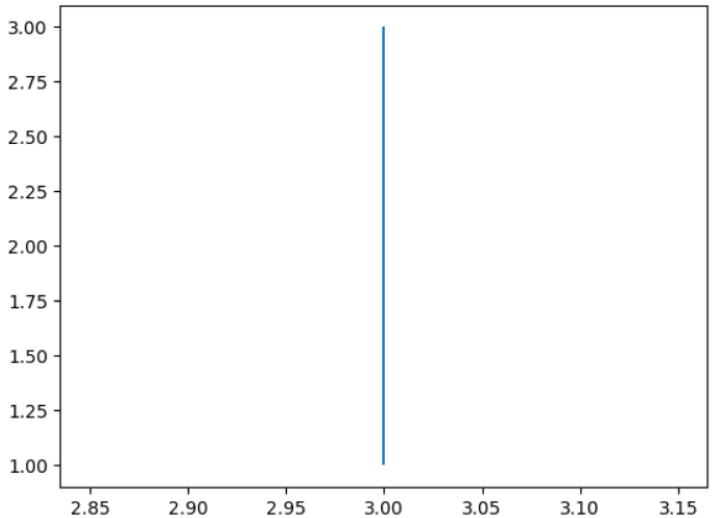
在上面的程式碼中,我們繪製了一條在 x 座標上的位置 3 以及在 y 座標上的 1 到 3 之間繪製的垂直線。
示例 2
在此示例中,我們將透過向 vlines() 傳遞其他引數(例如顏色和線寬)來自定義線的外觀。
import matplotlib.pyplot as plt plt.vlines(x = 2, ymin = 1, ymax = 3, color = 'green', linewidth = 3) plt.show()
輸出
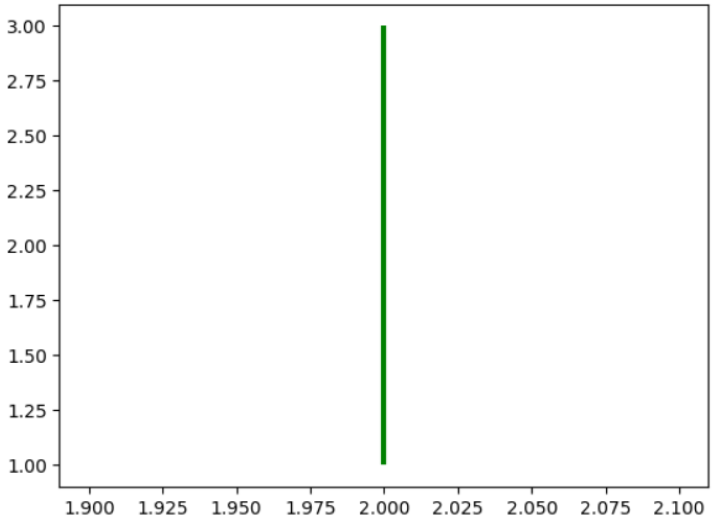
我們更改了先前示例的程式碼以繪製一條具有 3 的粗線寬的實心綠色線。
結論
在本文中,我們學習瞭如何使用 Matplotlib 繪製垂直線。我們已經看到了執行此操作的三種方法:使用 'axvline()'、'vlines()' 和 'plot()' 方法。根據我們的需求和偏好,每種方法都有其自身的優缺點。


 資料結構
資料結構 網路
網路 關係型資料庫管理系統 (RDBMS)
關係型資料庫管理系統 (RDBMS) 作業系統
作業系統 Java
Java iOS
iOS HTML
HTML CSS
CSS Android
Android Python
Python C 程式設計
C 程式設計 C++
C++ C#
C# MongoDB
MongoDB MySQL
MySQL Javascript
Javascript PHP
PHP